correlate2d#
- scipy.signal.correlate2d(in1, in2, mode='full', границе='fill', fillvalue=0)[источник]#
Взаимная корреляция двух двумерных массивов.
Взаимная корреляция in1 и in2 с размером выхода, определяемым mode, и граничные условия, определяемые границе и fillvalue.
- Параметры:
- in1array_like
Первый вход.
- in2array_like
Второй вход. Должен иметь то же количество измерений, что и in1.
- modestr {‘full’, ‘valid’, ‘same’}, необязательный
Строка, указывающая размер вывода:
fullВыходные данные представляют собой полную дискретную линейную взаимную корреляцию входных данных. (По умолчанию)
validВыходные данные состоят только из тех элементов, которые не зависят от дополнения нулями. В режиме 'valid' либо in1 или in2 должен быть как минимум таким же большим, как другой, по каждому измерению.
sameВыходные данные имеют тот же размер, что и in1, центрированный относительно вывода 'full'.
- границеstr {'fill', 'wrap', 'symm'}, опционально
Флаг, указывающий, как обрабатывать границы:
fillДополнить входные массивы значением fillvalue. (по умолчанию)
wrapциклические граничные условия.
symmсимметричные граничные условия.
- fillvalueскаляр, опционально
Значение для заполнения дополненных входных массивов. По умолчанию 0.
- Возвращает:
- correlate2dndarray
Двумерный массив, содержащий подмножество дискретной линейной кросс-корреляции in1 с in2.
Примечания
При использовании режима "same" с входами четной длины, выходы
correlateиcorrelate2dразличаются: между ними существует смещение на 1 индекс.Примеры
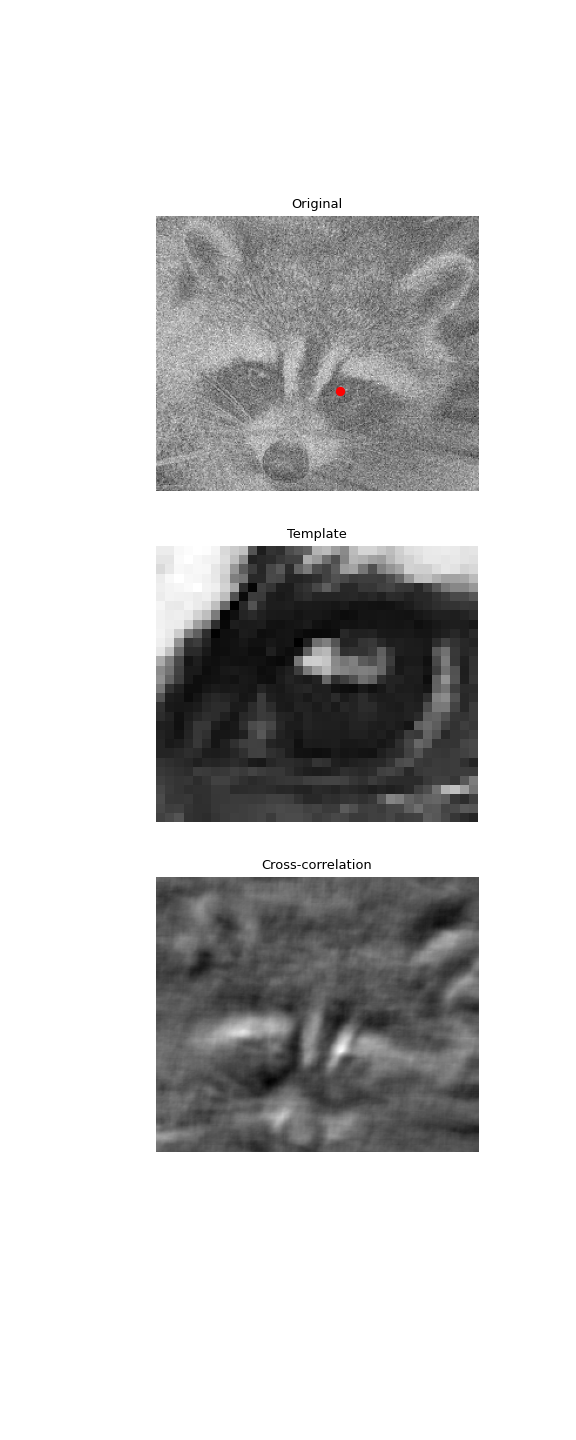
Использовать 2D кросс-корреляцию для нахождения местоположения шаблона в зашумлённом изображении:
>>> import numpy as np >>> from scipy import signal, datasets, ndimage >>> rng = np.random.default_rng() >>> face = datasets.face(gray=True) - datasets.face(gray=True).mean() >>> face = ndimage.zoom(face[30:500, 400:950], 0.5) # extract the face >>> template = np.copy(face[135:165, 140:175]) # right eye >>> template -= template.mean() >>> face = face + rng.standard_normal(face.shape) * 50 # add noise >>> corr = signal.correlate2d(face, template, boundary='symm', mode='same') >>> y, x = np.unravel_index(np.argmax(corr), corr.shape) # find the match
>>> import matplotlib.pyplot as plt >>> fig, (ax_orig, ax_template, ax_corr) = plt.subplots(3, 1, ... figsize=(6, 15)) >>> ax_orig.imshow(face, cmap='gray') >>> ax_orig.set_title('Original') >>> ax_orig.set_axis_off() >>> ax_template.imshow(template, cmap='gray') >>> ax_template.set_title('Template') >>> ax_template.set_axis_off() >>> ax_corr.imshow(corr, cmap='gray') >>> ax_corr.set_title('Cross-correlation') >>> ax_corr.set_axis_off() >>> ax_orig.plot(x, y, 'ro') >>> fig.show()