logrank#
- scipy.stats.logrank(x, y, альтернатива='two-sided')[источник]#
Сравнить распределения выживаемости двух выборок с помощью логранк-теста.
- Параметры:
- x, yarray_like или CensoredData
Выборки для сравнения на основе их эмпирических функций выживания.
- альтернатива{‘two-sided’, ‘less’, ‘greater’}, необязательный
Определяет альтернативную гипотезу.
Нулевая гипотеза состоит в том, что функции выживания двух групп, скажем X и Y, идентичны.
Следующие альтернативные гипотезы [4] доступны (по умолчанию «two-sided»):
'two-sided': функции выживания двух групп не идентичны.
‘less’: выживаемость группы X предпочтительнее: группа X функция интенсивности отказов меньше, чем группа Y функция интенсивности отказов в некоторые моменты времени.
‘greater’: выживание группы Y предпочтительнее: группа X функция интенсивности отказов больше, чем у группы Y функция интенсивности отказов в некоторые моменты времени.
- Возвращает:
- resLogRankResult
Объект, содержащий атрибуты:
- статистикаfloat ndarray
Вычисленная статистика (определена ниже). Её величина — это квадратный корень величины, возвращаемой большинством других реализаций теста логранга.
- p-значениеfloat ndarray
Вычисленное p-значение теста.
Смотрите также
Примечания
Тест логранга [1] сравнивает наблюдаемое количество событий с ожидаемым количеством событий при нулевой гипотезе, что две выборки были взяты из одного распределения. Статистика равна
\[Z_i = \frac{\sum_{j=1}^J(O_{i,j}-E_{i,j})}{\sqrt{\sum_{j=1}^J V_{i,j}}} \rightarrow \mathcal{N}(0,1)\]где
\[E_{i,j} = O_j \frac{N_{i,j}}{N_j}, \qquad V_{i,j} = E_{i,j} \left(\frac{N_j-O_j}{N_j}\right) \left(\frac{N_j-N_{i,j}}{N_j-1}\right),\]\(i\) обозначает группу (т.е. может принимать значения \(x\) или \(y\), или он может быть опущен для ссылки на объединённую выборку) \(j\) обозначает время (в которое произошло событие), \(N\) это количество субъектов в группе риска непосредственно перед наступлением события, и \(O\) это наблюдаемое количество событий в это время.
The
statistic\(Z_x\) возвращаемыйlogrank— это (знаковый) квадратный корень статистики, возвращаемой многими другими реализациями. При нулевой гипотезе \(Z_x**2\) асимптотически распределен согласно хи-квадрат распределению с одной степенью свободы. Следовательно, \(Z_x\) асимптотически распределена в соответствии со стандартным нормальным распределением. Преимущество использования \(Z_x\) сохраняется ли знак информации (т.е. имеет ли наблюдаемое количество событий тенденцию быть меньше или больше ожидаемого количества при нулевой гипотезе), что позволяетscipy.stats.logrankдля предложения односторонних альтернативных гипотез.Ссылки
[1]Мантел Н. "Оценка данных выживаемости и две новые статистики рангового порядка, возникающие при ее рассмотрении." Cancer Chemotherapy Reports, 50(3):163-170, PMID: 5910392, 1966
[2]Блэнд, Олтман, "Тест логранка", BMJ, 328:1073, DOI:10.1136/bmj.328.7447.1073, 2004
[3]“Logrank test”, Википедия, https://en.wikipedia.org/wiki/Logrank_test
[4]Brown, Mark. “On the choice of variance for the log rank test.” Biometrika 71.1 (1984): 65-74.
[5]Клайн, Джон П., и Мелвин Л. Мошбергер. Анализ выживаемости: методы для цензурированных и усечённых данных. Том 1230. Нью-Йорк: Springer, 2003.
Примеры
Ссылка [2] сравнил время выживания пациентов с двумя разными типами рецидивирующих злокачественных глиом. Образцы ниже записывают время (количество недель), в течение которого каждый пациент участвовал в исследовании.
scipy.stats.CensoredDataкласс используется, потому что данные являются правоцензурированными: нецензурированные наблюдения соответствуют наблюдаемым смертям, тогда как цензурированные наблюдения соответствуют случаям, когда пациент покинул исследование по другой причине.>>> from scipy import stats >>> x = stats.CensoredData( ... uncensored=[6, 13, 21, 30, 37, 38, 49, 50, ... 63, 79, 86, 98, 202, 219], ... right=[31, 47, 80, 82, 82, 149] ... ) >>> y = stats.CensoredData( ... uncensored=[10, 10, 12, 13, 14, 15, 16, 17, 18, 20, 24, 24, ... 25, 28,30, 33, 35, 37, 40, 40, 46, 48, 76, 81, ... 82, 91, 112, 181], ... right=[34, 40, 70] ... )
Мы можем вычислить и визуализировать эмпирические функции выживания обеих групп следующим образом.
>>> import numpy as np >>> import matplotlib.pyplot as plt >>> ax = plt.subplot() >>> ecdf_x = stats.ecdf(x) >>> ecdf_x.sf.plot(ax, label='Astrocytoma') >>> ecdf_y = stats.ecdf(y) >>> ecdf_y.sf.plot(ax, label='Glioblastoma') >>> ax.set_xlabel('Time to death (weeks)') >>> ax.set_ylabel('Empirical SF') >>> plt.legend() >>> plt.show()
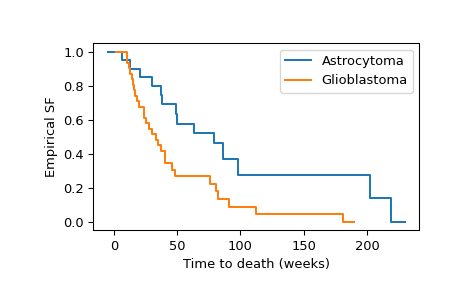
Визуальный анализ эмпирических функций выживания предполагает, что времена выживания имеют тенденцию различаться между двумя группами. Для формальной оценки значимости различия на уровне 1% используем логарифмический ранговый критерий.
>>> res = stats.logrank(x=x, y=y) >>> res.statistic -2.73799 >>> res.pvalue 0.00618
P-значение меньше 1%, поэтому мы можем считать данные свидетельством против нулевой гипотезы в пользу альтернативы о наличии разницы между двумя функциями выживаемости.