Коэффициент корреляции Спирмена#
Коэффициент корреляции Спирмена — это непараметрическая мера монотонности связи между двумя наборами данных.
Рассмотрим следующие данные из [1], который изучал взаимосвязь между свободным пролином (аминокислотой) и общим коллагеном (белком, часто встречающимся в соединительной ткани) в больной человеческой печени.
The x и y массивы ниже записывают измерения двух соединений.
Наблюдения парные: каждое измерение свободного пролина было взято из той же
печени, что и измерение общего коллагена с тем же индексом.
import numpy as np
# total collagen (mg/g dry weight of liver)
x = np.array([7.1, 7.1, 7.2, 8.3, 9.4, 10.5, 11.4])
# free proline (μ mole/g dry weight of liver)
y = np.array([2.8, 2.9, 2.8, 2.6, 3.5, 4.6, 5.0])
Эти данные были проанализированы в [2] используя коэффициент корреляции Спирмена,
статистику, чувствительную к монотонной корреляции между выборками, реализованную
как scipy.stats.spearmanr.
from scipy import stats
res = stats.spearmanr(x, y)
res.statistic
np.float64(0.7000000000000001)
Значение этой статистики стремится быть высоким (близким к 1) для выборок с сильно положительной порядковой корреляцией, низким (близким к -1) для выборок с сильно отрицательной порядковой корреляцией и малым по величине (близким к нулю) для выборок со слабой порядковой корреляцией.
Тест выполняется путем сравнения наблюдаемого значения статистики с нулевым распределением: распределением значений статистики, полученных при нулевой гипотезе о том, что измерения общего коллагена и свободного пролина независимы.
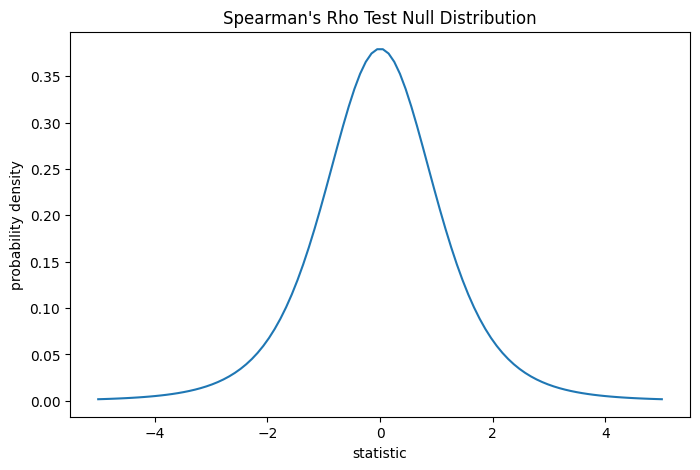
Для этого теста статистика может быть преобразована таким образом, что нулевое распределение для больших выборок является t-распределением Стьюдента с len(x) - 2 степени свободы.
import matplotlib.pyplot as plt
dof = len(x)-2 # len(x) == len(y)
dist = stats.t(df=dof)
t_vals = np.linspace(-5, 5, 100)
pdf = dist.pdf(t_vals)
fig, ax = plt.subplots(figsize=(8, 5))
def plot(ax): # we'll reuse this
ax.plot(t_vals, pdf)
ax.set_title("Spearman's Rho Test Null Distribution")
ax.set_xlabel("statistic")
ax.set_ylabel("probability density")
plot(ax)
plt.show()
Сравнение количественно оценивается p-значением: долей значений в нулевом распределении, столь же экстремальных или более экстремальных, чем наблюдаемое значение статистики. В двустороннем тесте, в котором статистика положительна, элементы нулевого распределения больше преобразованной статистики и элементы нулевого распределения меньше отрицательного наблюдаемой статистики оба считаются "более экстремальными".
fig, ax = plt.subplots(figsize=(8, 5))
plot(ax)
rs = res.statistic # original statistic
transformed = rs * np.sqrt(dof / ((rs+1.0)*(1.0-rs)))
pvalue = dist.cdf(-transformed) + dist.sf(transformed)
annotation = (f'p-value={pvalue:.4f}\n(shaded area)')
props = dict(facecolor='black', width=1, headwidth=5, headlength=8)
_ = ax.annotate(annotation, (2.7, 0.025), (3, 0.03), arrowprops=props)
i = t_vals >= transformed
ax.fill_between(t_vals[i], y1=0, y2=pdf[i], color='C0')
i = t_vals <= -transformed
ax.fill_between(t_vals[i], y1=0, y2=pdf[i], color='C0')
ax.set_xlim(-5, 5)
ax.set_ylim(0, 0.1)
plt.show()
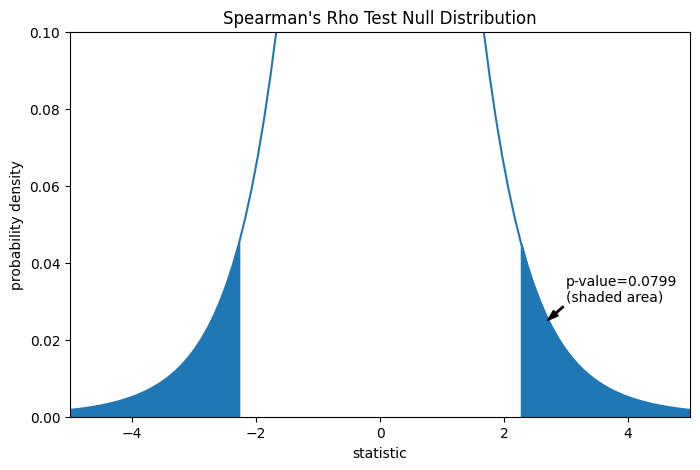
res.pvalue
np.float64(0.07991669030889909)
Если p-значение «маленькое» — то есть если существует низкая вероятность выборки данных из независимых распределений, которые дают такое экстремальное значение статистики — это может рассматриваться как свидетельство против нулевой гипотезы в пользу альтернативной: распределение общего коллагена и свободного пролина не независимы. Обратите внимание, что:
Обратное неверно; то есть тест не используется для предоставления доказательств в пользу нулевой гипотезы.
Порог для значений, которые будут считаться «малыми», — это выбор, который следует сделать до анализа данных [3] с учетом рисков как ложноположительных (ошибочное отклонение нулевой гипотезы), так и ложноотрицательных (неспособность отклонить ложную нулевую гипотезу).
Маленькие p-значения не являются доказательством большой эффект; скорее, они могут только предоставить доказательства «значимого» эффекта, означающего, что они маловероятны при нулевой гипотезе.
Предположим, что до проведения эксперимента у авторов были основания предсказывать положительную корреляцию между измерениями общего коллагена и свободного пролина, и они выбрали оценку правдоподобия нулевой гипотезы против односторонней альтернативы: свободный пролин имеет положительную порядковую корреляцию с общим коллагеном. В этом случае только те значения в нулевом распределении, которые равны или больше наблюдаемой статистики, считаются более экстремальными.
res = stats.spearmanr(x, y, alternative='greater')
res.statistic
np.float64(0.7000000000000001)
fig, ax = plt.subplots(figsize=(8, 5))
plot(ax)
pvalue = dist.sf(transformed)
annotation = (f'p-value={pvalue:.6f}\n(shaded area)')
props = dict(facecolor='black', width=1, headwidth=5, headlength=8)
_ = ax.annotate(annotation, (3, 0.018), (3.5, 0.03), arrowprops=props)
i = t_vals >= transformed
ax.fill_between(t_vals[i], y1=0, y2=pdf[i], color='C0')
ax.set_xlim(1, 5)
ax.set_ylim(0, 0.1)
plt.show()
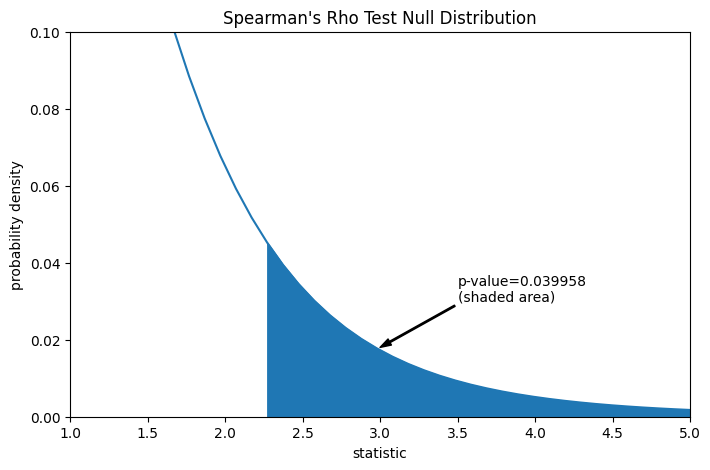
res.pvalue
np.float64(0.03995834515444954)
Обратите внимание, что t-распределение предоставляет асимптотическое приближение нулевого
распределения; оно точно только для выборок с большим количеством наблюдений. Для малых
выборок может быть более уместно выполнить перестановочный тест [4]: В соответствии с
нулевой гипотезой о независимости общего коллагена и свободного пролина, каждое из
измерений свободного пролина было одинаково вероятно наблюдаться с любым
из измерений общего коллагена. Поэтому мы можем сформировать точный нулевое распределение путем вычисления статистики для каждой возможной пары элементов между x и y.
def statistic(x): # explore all possible pairings by permuting `x`
rs = stats.spearmanr(x, y).statistic # ignore pvalue
transformed = rs * np.sqrt(dof / ((rs+1.0)*(1.0-rs)))
return transformed
ref = stats.permutation_test((x,), statistic, alternative='greater',
permutation_type='pairings')
fig, ax = plt.subplots(figsize=(8, 5))
plot(ax)
ax.hist(ref.null_distribution, np.linspace(-5, 5, 26),
density=True)
ax.legend(['asymptotic approximation\n(many observations)',
f'exact \n({len(ref.null_distribution)} permutations)'])
plt.show()
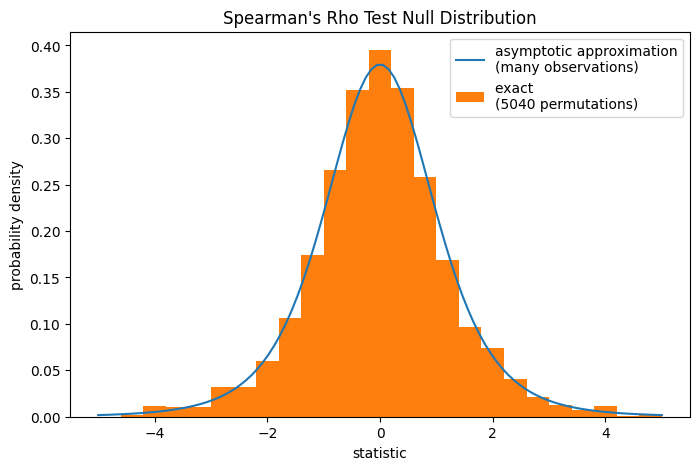
ref.pvalue
np.float64(0.04563492063492063)