OPTICS#
- класс sklearn.cluster.OPTICS(*, min_samples=5, max_eps=inf, метрика='minkowski', p=2, metric_params=None, cluster_method='xi', eps=None, xi=0.05, предшествующая_коррекция=True, min_cluster_size=None, алгоритм='auto', leaf_size=30, память=None, n_jobs=None)[источник]#
Оценка структуры кластеризации из массива векторов.
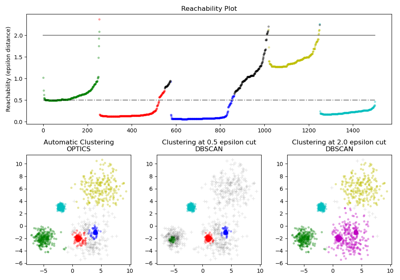
OPTICS (Ordering Points To Identify the Clustering Structure), тесно связанный с DBSCAN, находит ядерные образцы высокой плотности и расширяет кластеры из них [1]. В отличие от DBSCAN, он сохраняет иерархию кластеров для переменного радиуса окрестности. Лучше подходит для использования на больших наборах данных, чем текущая реализация DBSCAN в scikit-learn.
Кластеры затем извлекаются из порядка кластеров с использованием метода, подобного DBSCAN (cluster_method = 'dbscan'), или автоматической техники, предложенной в [1] (cluster_method = ‘xi’).
Эта реализация отклоняется от оригинального OPTICS, сначала выполняя поиск k-ближайших соседей по всем точкам для определения размеров ядра всех точек (вместо вычисления соседей во время цикла по точкам). Затем вычисляются расстояния достижимости только для необработанных точек, чтобы построить порядок кластеризации, аналогичный оригинальному OPTICS. Обратите внимание, что мы не используем кучу для управления кандидатами на расширение, поэтому временная сложность будет O(n^2).
Подробнее в Руководство пользователя.
- Параметры:
- min_samplesint > 1 или float между 0 и 1, по умолчанию=5
Количество образцов в окрестности точки, чтобы она считалась центральной точкой. Также, восходящие и нисходящие крутые области не могут иметь более
min_samplesпоследовательные не крутые точки. Выражается как абсолютное число или доля от количества выборок (округляется до не менее 2).- max_epsfloat, по умолчанию=np.inf
Максимальное расстояние между двумя образцами, чтобы один считался в окрестности другого. Значение по умолчанию
np.infбудет идентифицировать кластеры на всех масштабах; уменьшениеmax_epsприведет к более короткому времени выполнения.- метрикаstr или callable, по умолчанию='minkowski'
Метрика для вычисления расстояния. Любая метрика из scikit-learn или
scipy.spatial.distanceможет быть использован.Если
metricявляется вызываемой функцией, она вызывается для каждой пары экземпляров (строк), и полученное значение записывается. Вызываемая функция должна принимать два массива на вход и возвращать одно значение, указывающее расстояние между ними. Это работает для метрик Scipy, но менее эффективно, чем передача имени метрики в виде строки. Если метрика «precomputed»,Xпредполагается матрицей расстояний и должна быть квадратной.Допустимые значения для metric:
из scikit-learn: ['cityblock', 'cosine', 'euclidean', 'l1', 'l2', 'manhattan']
из scipy.spatial.distance: [‘braycurtis’, ‘canberra’, ‘chebyshev’, ‘correlation’, ‘dice’, ‘hamming’, ‘jaccard’, ‘kulsinski’, ‘mahalanobis’, ‘minkowski’, ‘rogerstanimoto’, ‘russellrao’, ‘seuclidean’, ‘sokalmichener’, ‘sokalsneath’, ‘sqeuclidean’, ‘yule’]
Разреженные матрицы поддерживаются только метриками scikit-learn. См.
scipy.spatial.distanceдля подробностей об этих метриках.Примечание
'kulsinski'устарел в SciPy 1.9 и будет удален в SciPy 1.11.- pfloat, по умолчанию=2
Параметр для метрики Минковского из
pairwise_distances. При p = 1 это эквивалентно использованию manhattan_distance (l1), а euclidean_distance (l2) для p = 2. Для произвольного p используется minkowski_distance (l_p).- metric_paramsdict, по умолчанию=None
Дополнительные именованные аргументы для метрической функции.
- cluster_method{‘xi’, ‘dbscan’}, по умолчанию ‘xi’
Метод извлечения, используемый для извлечения кластеров с использованием рассчитанной достижимости и упорядочения.
- epsfloat, по умолчанию=None
Максимальное расстояние между двумя образцами, чтобы один считался в окрестности другого. По умолчанию принимает то же значение, что и
max_eps. Используется только когдаcluster_method='dbscan'.- xiчисло с плавающей точкой от 0 до 1, по умолчанию=0.05
Определяет минимальную крутизну на графике достижимости, которая образует границу кластера. Например, восходящая точка на графике достижимости определяется соотношением от одной точки к её преемнику, составляющим не более 1-xi. Используется только когда
cluster_method='xi'.- предшествующая_коррекцияbool, по умолчанию=True
Корректные кластеры согласно предшественникам, рассчитанным OPTICS [2]. Этот параметр оказывает минимальное влияние на большинство наборов данных. Используется только когда
cluster_method='xi'.- min_cluster_sizeint > 1 или float между 0 и 1, по умолчанию=None
Минимальное количество образцов в кластере OPTICS, выраженное как абсолютное число или доля от общего числа образцов (округляется до значения не менее 2). Если
None, значениеmin_samplesиспользуется вместо. Используется только когдаcluster_method='xi'.- алгоритм{‘auto’, ‘ball_tree’, ‘kd_tree’, ‘brute’}, по умолчанию=’auto’
Алгоритм, используемый для вычисления ближайших соседей:
'ball_tree' будет использовать
BallTree.'kd_tree' будет использовать
KDTree.'brute' будет использовать поиск методом грубой силы.
‘auto’ (по умолчанию) попытается определить наиболее подходящий алгоритм на основе значений, переданных в
fitметод.
Примечание: обучение на разреженных входных данных переопределит настройку этого параметра, используя метод грубой силы.
- leaf_sizeint, по умолчанию=30
Размер листа, передаваемый в
BallTreeилиKDTree. Это может повлиять на скорость построения и запроса, а также на память, необходимую для хранения дерева. Оптимальное значение зависит от характера задачи.- памятьstr или объект с интерфейсом joblib.Memory, по умолчанию=None
Используется для кэширования результатов вычисления дерева. По умолчанию кэширование не выполняется. Если указана строка, это путь к директории кэширования.
- n_jobsint, default=None
Количество параллельных задач для поиска соседей.
Noneозначает 1, если только не вjoblib.parallel_backendконтекст.-1означает использование всех процессоров. См. Глоссарий для получения дополнительной информации.
- Атрибуты:
- labels_ndarray формы (n_samples,)
Метки кластеров для каждой точки в наборе данных, переданном в fit(). Шумные образцы и точки, не включённые в листовой кластер
cluster_hierarchy_помечены как -1.- reachability_ndarray формы (n_samples,)
Расстояния достижимости для каждого образца, индексированные по порядку объектов. Используйте
clust.reachability_[clust.ordering_]для доступа в порядке кластера.- ordering_ndarray формы (n_samples,)
Упорядоченный список индексов образцов для кластера.
- core_distances_ndarray формы (n_samples,)
Расстояние, на котором каждый образец становится центральной точкой, индексированное по порядку объектов. Точки, которые никогда не будут центральными, имеют расстояние inf. Используйте
clust.core_distances_[clust.ordering_]для доступа в порядке кластера.- предшественник_ndarray формы (n_samples,)
Точка, из которой была достигнута выборка, индексируется по порядку объектов. Начальные точки имеют предшественника -1.
- cluster_hierarchy_ndarray формы (n_clusters, 2)
Список кластеров в виде
[start, end]в каждой строке, с всеми индексами включительно. Кластеры упорядочены в соответствии с(end, -start)(по возрастанию), так что большие кластеры, включающие меньшие кластеры, идут после этих меньших. Посколькуlabels_не отражает иерархию, обычноlen(cluster_hierarchy_) > np.unique(optics.labels_). Пожалуйста, также обратите внимание, что эти индексы относятся кordering_, т.е.X[ordering_][start:end + 1]образуют кластер. Доступно только, когдаcluster_method='xi'.- n_features_in_int
Количество признаков, замеченных во время fit.
Добавлено в версии 0.24.
- feature_names_in_ndarray формы (
n_features_in_,) Имена признаков, наблюдаемых во время fit. Определено только когда
Xимеет имена признаков, которые все являются строками.Добавлено в версии 1.0.
Смотрите также
DBSCANАналогичная кластеризация для заданного радиуса окрестности (eps). Наша реализация оптимизирована для времени выполнения.
Ссылки
[1] (1,2)Анкерст, Михаэль, Маркус М. Бройниг, Ханс-Петер Кригель и Йорг Зандер. "OPTICS: упорядочивание точек для идентификации структуры кластеризации." ACM SIGMOD Record 28, № 2 (1999): 49-60.
[2]Шуберт, Эрих, Майкл Герц. “Улучшение структуры кластеров, извлеченной из графиков OPTICS.” Proc. of the Conference “Lernen, Wissen, Daten, Analysen” (LWDA) (2018): 318-329.
Примеры
>>> from sklearn.cluster import OPTICS >>> import numpy as np >>> X = np.array([[1, 2], [2, 5], [3, 6], ... [8, 7], [8, 8], [7, 3]]) >>> clustering = OPTICS(min_samples=2).fit(X) >>> clustering.labels_ array([0, 0, 0, 1, 1, 1])
Более подробный пример см. в Демонстрация алгоритма кластеризации OPTICS.
Для сравнения OPTICS с другими алгоритмами кластеризации см. Сравнение различных алгоритмов кластеризации на игрушечных наборах данных
- fit(X, y=None)[источник]#
Выполнение кластеризации OPTICS.
Извлекает упорядоченный список точек и расстояний достижимости, а также выполняет начальную кластеризацию с использованием
max_epsрасстояние, указанное при создании объекта OPTICS.- Параметры:
- X{ndarray, разреженная матрица} формы (n_samples, n_features), или (n_samples, n_samples) если metric='precomputed'
Массив признаков или массив расстояний между образцами, если metric='precomputed'. Если предоставлена разреженная матрица, она будет преобразована в формат CSR.
- yИгнорируется
Не используется, присутствует для согласованности API по соглашению.
- Возвращает:
- selfobject
Возвращает обученный экземпляр self.
- fit_predict(X, y=None, **kwargs)[источник]#
Выполнить кластеризацию на
Xи возвращает метки кластеров.- Параметры:
- Xarray-like формы (n_samples, n_features)
Входные данные.
- yИгнорируется
Не используется, присутствует для согласованности API по соглашению.
- **kwargsdict
Аргументы, передаваемые в
fit.Добавлено в версии 1.4.
- Возвращает:
- меткиndarray формы (n_samples,), dtype=np.int64
Метки кластеров.
- 6332()[источник]#
Получить маршрутизацию метаданных этого объекта.
Пожалуйста, проверьте Руководство пользователя о том, как работает механизм маршрутизации.
- Возвращает:
- маршрутизацияMetadataRequest
A
MetadataRequestИнкапсуляция информации о маршрутизации.
- get_params(глубокий=True)[источник]#
Получить параметры для этого оценщика.
- Параметры:
- глубокийbool, по умолчанию=True
Если True, вернет параметры для этого оценщика и вложенных подобъектов, которые являются оценщиками.
- Возвращает:
- paramsdict
Имена параметров, сопоставленные с их значениями.
- set_params(**params)[источник]#
Установить параметры этого оценщика.
Метод работает как на простых оценщиках, так и на вложенных объектах (таких как
Pipeline). Последние имеют параметры вида__ - Параметры:
- **paramsdict
Параметры оценщика.
- Возвращает:
- selfэкземпляр estimator
Экземпляр оценщика.
Примеры галереи#
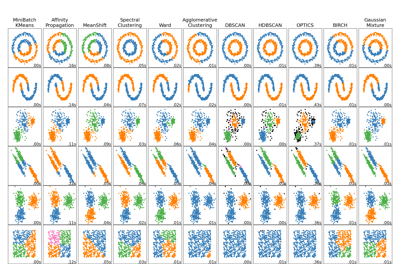
Сравнение различных алгоритмов кластеризации на игрушечных наборах данных