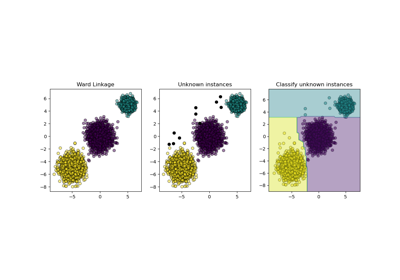
AgglomerativeClustering#
- класс sklearn.cluster.AgglomerativeClustering(n_clusters=2, *, метрика='euclidean', память=None, связность=None, compute_full_tree='auto', linkage='ward', distance_threshold=None, compute_distances=False)[источник]#
Агломеративная кластеризация.
Рекурсивно объединяет пары кластеров выборочных данных; использует расстояние связи.
Подробнее в Руководство пользователя.
- Параметры:
- n_clustersint или None, по умолчанию=2
Количество кластеров для поиска. Оно должно быть
Noneifdistance_thresholdне являетсяNone.- метрикаstr или callable, по умолчанию="euclidean"
Метрика, используемая для вычисления связи. Может быть "euclidean", "l1", "l2", "manhattan", "cosine" или "precomputed". Если связь "ward", принимается только "euclidean". Если "precomputed", для метода fit требуется матрица расстояний. Если связность None, связь "single" и сходство не "precomputed", можно назначить любую допустимую метрику попарного расстояния.
Пример агломеративной кластеризации с различными метриками см. в Агломеративная кластеризация с разными метриками.
Добавлено в версии 1.2.
- памятьstr или объект с интерфейсом joblib.Memory, по умолчанию=None
Используется для кэширования результатов вычисления дерева. По умолчанию кэширование не выполняется. Если указана строка, это путь к директории кэширования.
- связностьarray-like, sparse matrix, или callable, по умолчанию=None
Матрица связности. Определяет для каждого образца соседние образцы в соответствии с заданной структурой данных. Это может быть сама матрица связности или вызываемый объект, который преобразует данные в матрицу связности, например, полученную из
kneighbors_graph. По умолчаниюNone, т.е., алгоритм иерархической кластеризации неструктурирован.Для примера матрицы связности с использованием
kneighbors_graph, см. Иерархическая кластеризация со структурой и без.- compute_full_tree‘auto’ или bool, по умолчанию=’auto’
Остановить раннее построение дерева на
n_clusters. Это полезно для уменьшения времени вычислений, если количество кластеров не мало по сравнению с количеством выборок. Эта опция полезна только при указании матрицы связности. Также обратите внимание, что при изменении количества кластеров и использовании кэширования может быть выгодно вычислять полное дерево. Оно должно бытьTrueifdistance_thresholdне являетсяNone. По умолчаниюcompute_full_treeравно “auto”, что эквивалентноTrueкогдаdistance_thresholdне являетсяNoneили чтоn_clustersхуже максимума между 100 или0.02 * n_samples. В противном случае, “auto” эквивалентноFalse.- linkage{'ward', 'complete', 'average', 'single'}, по умолчанию='ward'
Какой критерий связи использовать. Критерий связи определяет, какое расстояние использовать между наборами наблюдений. Алгоритм будет объединять пары кластеров, которые минимизируют этот критерий.
‘ward’ минимизирует дисперсию объединяемых кластеров.
'average' использует среднее расстояний каждого наблюдения двух наборов.
Связь 'complete' или 'maximum' использует максимальные расстояния между всеми наблюдениями двух наборов.
'single' использует минимум расстояний между всеми наблюдениями двух наборов.
Добавлено в версии 0.20: Добавлена опция 'single'
Для примеров, сравнивающих различные
linkageкритерии, см. Сравнение различных методов иерархической связи на игрушечных наборах данных.- distance_thresholdfloat, по умолчанию=None
Пороговое значение расстояния связи, при котором или выше которого кластеры не будут объединяться. Если не
None,n_clustersдолжен бытьNoneиcompute_full_treeдолжен бытьTrue.Добавлено в версии 0.21.
- compute_distancesbool, по умолчанию=False
Вычисляет расстояния между кластерами, даже если
distance_thresholdне используется. Это может быть использовано для визуализации дендрограммы, но влечет за собой вычислительные и ресурсные затраты.Добавлено в версии 0.24.
Для примера визуализации дендрограммы см. Построить дендрограмму иерархической кластеризации.
- Атрибуты:
- n_clusters_int
Количество кластеров, найденных алгоритмом. Если
distance_threshold=None, он будет равен заданномуn_clusters.- labels_ndarray формы (n_samples)
Метки кластеров для каждой точки.
- n_leaves_int
Количество листьев в иерархическом дереве.
- n_connected_components_int
Оценочное количество связных компонент в графе.
Добавлено в версии 0.21:
n_connected_components_был добавлен для заменыn_components_.- n_features_in_int
Количество признаков, замеченных во время fit.
Добавлено в версии 0.24.
- feature_names_in_ndarray формы (
n_features_in_,) Имена признаков, наблюдаемых во время fit. Определено только когда
Xимеет имена признаков, которые все являются строками.Добавлено в версии 1.0.
- children_массивоподобный формы (n_samples-1, 2)
Дочерние элементы каждого нелистового узла. Значения меньше
n_samplesсоответствуют листьям дерева, которые являются исходными образцами. Узелiбольше или равноn_samplesявляется нелистовым узлом и имеет дочерние узлыchildren_[i - n_samples]. Альтернативно, на i-й итерации children[i][0] и children[i][1] объединяются, чтобы сформировать узелn_samples + i.- distances_массивоподобный формы (n_nodes-1,)
Расстояния между узлами в соответствующем месте в
children_. Вычисляется только еслиdistance_thresholdиспользуется илиcompute_distancesустановлено вTrue.
Смотрите также
FeatureAgglomerationАгломеративная кластеризация, но для признаков вместо образцов.
ward_treeИерархическая кластеризация с методом связывания Уорда.
Примеры
>>> from sklearn.cluster import AgglomerativeClustering >>> import numpy as np >>> X = np.array([[1, 2], [1, 4], [1, 0], ... [4, 2], [4, 4], [4, 0]]) >>> clustering = AgglomerativeClustering().fit(X) >>> clustering AgglomerativeClustering() >>> clustering.labels_ array([1, 1, 1, 0, 0, 0])
Для сравнения агломеративной кластеризации с другими алгоритмами кластеризации см. Сравнение различных алгоритмов кластеризации на игрушечных наборах данных
- fit(X, y=None)[источник]#
Обучить иерархическую кластеризацию по признакам или матрице расстояний.
- Параметры:
- Xarray-like, shape (n_samples, n_features) или (n_samples, n_samples)
Обучающие экземпляры для кластеризации, или расстояния между экземплярами, если
metric='precomputed'.- yИгнорируется
Не используется, присутствует здесь для согласованности API по соглашению.
- Возвращает:
- selfobject
Возвращает обученный экземпляр.
- fit_predict(X, y=None)[источник]#
Обучить и вернуть результат кластеризации для каждого образца.
В дополнение к обучению, этот метод также возвращает результат кластеризации для каждого образца в обучающем наборе.
- Параметры:
- Xмассивоподобный объект формы (n_samples, n_features) или (n_samples, n_samples)
Обучающие экземпляры для кластеризации, или расстояния между экземплярами, если
affinity='precomputed'.- yИгнорируется
Не используется, присутствует здесь для согласованности API по соглашению.
- Возвращает:
- меткиndarray формы (n_samples,)
Метки кластеров.
- 6332()[источник]#
Получить маршрутизацию метаданных этого объекта.
Пожалуйста, проверьте Руководство пользователя о том, как работает механизм маршрутизации.
- Возвращает:
- маршрутизацияMetadataRequest
A
MetadataRequestИнкапсуляция информации о маршрутизации.
- get_params(глубокий=True)[источник]#
Получить параметры для этого оценщика.
- Параметры:
- глубокийbool, по умолчанию=True
Если True, вернет параметры для этого оценщика и вложенных подобъектов, которые являются оценщиками.
- Возвращает:
- paramsdict
Имена параметров, сопоставленные с их значениями.
- set_params(**params)[источник]#
Установить параметры этого оценщика.
Метод работает как на простых оценщиках, так и на вложенных объектах (таких как
Pipeline). Последние имеют параметры вида__ - Параметры:
- **paramsdict
Параметры оценщика.
- Возвращает:
- selfэкземпляр estimator
Экземпляр оценщика.
Примеры галереи#
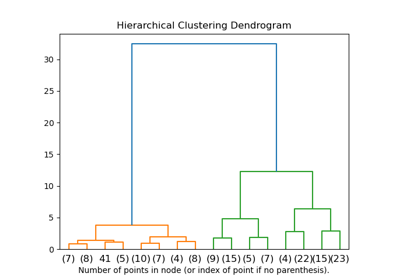
Построить дендрограмму иерархической кластеризации
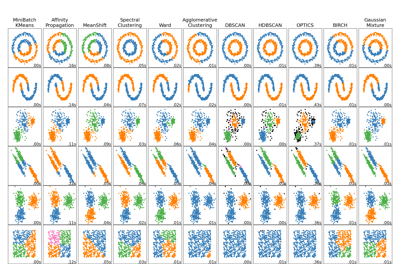
Сравнение различных алгоритмов кластеризации на игрушечных наборах данных

Демонстрация структурированной иерархической кластеризации Уорда на изображении монет
Различные агломеративные кластеризации на 2D-вложении цифр
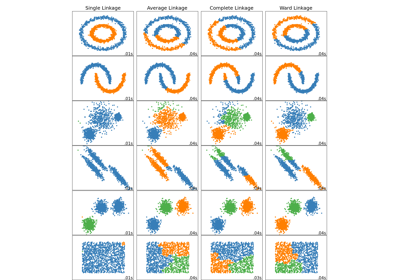
Сравнение различных методов иерархической связи на игрушечных наборах данных