DBSCAN#
- класс sklearn.cluster.DBSCAN(eps=0.5, *, min_samples=5, метрика='euclidean', metric_params=None, алгоритм='auto', leaf_size=30, p=None, n_jobs=None)[источник]#
Выполнить кластеризацию DBSCAN из массива векторов или матрицы расстояний.
DBSCAN - пространственная кластеризация приложений с шумом на основе плотности. Находит ядровые выборки высокой плотности и расширяет кластеры из них. Этот алгоритм особенно хорош для данных, содержащих кластеры схожей плотности, и может находить кластеры произвольной формы.
В отличие от K-средних, DBSCAN не требует указания количества кластеров заранее и может идентифицировать выбросы как шумовые точки.
Эта реализация имеет наихудшую сложность по памяти \(O({n}^2)\), что может произойти, когда
epsпараметр велик иmin_samplesнизко, в то время как оригинальный DBSCAN использует только линейную память. Для получения дополнительных сведений см. примечания ниже.Подробнее в Руководство пользователя.
- Параметры:
- epsfloat, по умолчанию=0.5
Максимальное расстояние между двумя образцами, чтобы один считался в окрестности другого. Это не максимальная граница на расстояниях точек внутри кластера. Это самый важный параметр DBSCAN для правильного выбора для вашего набора данных и функции расстояния. Меньшие значения обычно приводят к большему количеству кластеров.
- min_samplesint, по умолчанию=5
Количество образцов (или общий вес) в окрестности точки, чтобы она считалась основной точкой. Это включает саму точку. Если
min_samplesустановлено в более высокое значение, DBSCAN найдет более плотные кластеры, тогда как если оно установлено в более низкое значение, найденные кластеры будут более разреженными.- метрикаstr, или вызываемый объект, по умолчанию='euclidean'
Метрика для использования при вычислении расстояния между экземплярами в массиве признаков. Если метрика является строкой или вызываемым объектом, она должна быть одной из допустимых опций, разрешенных
sklearn.metrics.pairwise_distancesдля его параметра metric. Если metric равен "precomputed", предполагается, что X является матрицей расстояний и должен быть квадратным. X может быть разреженный граф, в этом случае только «ненулевые» элементы могут считаться соседями для DBSCAN.Добавлено в версии 0.17: метрика precomputed для приема предварительно вычисленной разреженной матрицы.
- metric_paramsdict, по умолчанию=None
Дополнительные именованные аргументы для метрической функции.
Добавлено в версии 0.19.
- алгоритм{‘auto’, ‘ball_tree’, ‘kd_tree’, ‘brute’}, по умолчанию=’auto’
Алгоритм, используемый модулем NearestNeighbors для вычисления попарных расстояний и поиска ближайших соседей. 'auto' попытается определить наиболее подходящий алгоритм на основе значений, переданных в
fitметод. См.NearestNeighborsдокументации для подробностей.- leaf_sizeint, по умолчанию=30
Размер листа, передаваемый в BallTree или cKDTree. Это может повлиять на скорость построения и запроса, а также на память, необходимую для хранения дерева. Оптимальное значение зависит от характера задачи.
- pfloat, по умолчанию=None
Степень метрики Минковского, используемая для вычисления расстояния между точками. Если None, то
p=2(эквивалентно евклидовому расстоянию). При p=1 это эквивалентно расстоянию Манхэттена.- n_jobsint, default=None
Количество параллельных задач для выполнения.
Noneозначает 1, если только не вjoblib.parallel_backendконтекст.-1означает использование всех процессоров. См. Глоссарий для получения дополнительной информации.
- Атрибуты:
- core_sample_indices_ndarray формы (n_core_samples,)
Индексы основных выборок.
- components_ndarray формы (n_core_samples, n_features)
Копия каждого основного образца, найденного при обучении.
- labels_ndarray формы (n_samples,)
Метки кластеров для каждой точки в наборе данных, переданном в fit(). Шумные выборки получают метку -1. Неотрицательные целые числа указывают принадлежность к кластеру.
- n_features_in_int
Количество признаков, замеченных во время fit.
Добавлено в версии 0.24.
- feature_names_in_ndarray формы (
n_features_in_,) Имена признаков, наблюдаемых во время fit. Определено только когда
Xимеет имена признаков, которые все являются строками.Добавлено в версии 1.0.
Смотрите также
OPTICSПохожая кластеризация при нескольких значениях eps. Наша реализация оптимизирована для использования памяти.
Примечания
Эта реализация массово вычисляет все запросы соседства, что увеличивает сложность по памяти до O(n.d), где d — среднее количество соседей, в то время как исходный DBSCAN имел сложность по памяти O(n). Это может привести к более высокой сложности по памяти при запросе этих ближайших окрестностей, в зависимости от
algorithm.Один из способов избежать сложности запросов — предварительно вычислять разреженные окрестности блоками с помощью
NearestNeighbors.radius_neighbors_graphсmode='distance'затем используяmetric='precomputed'здесь.Другой способ уменьшить использование памяти и времени вычислений — удалить (почти)дублирующиеся точки и использовать
sample_weightвместо этого.OPTICSпредоставляет аналогичную кластеризацию с меньшим использованием памяти.Ссылки
Ester, M., H. P. Kriegel, J. Sander, and X. Xu, “A Density-Based Algorithm for Discovering Clusters in Large Spatial Databases with Noise”для примера использования матрицы ошибок для классификации текстовых документов.
Schubert, E., Sander, J., Ester, M., Kriegel, H. P., & Xu, X. (2017). «DBSCAN пересмотрен, пересмотрен: почему и как вам (всё ещё) следует использовать DBSCAN.» ACM Transactions on Database Systems (TODS), 42(3), 19.
Примеры
>>> from sklearn.cluster import DBSCAN >>> import numpy as np >>> X = np.array([[1, 2], [2, 2], [2, 3], ... [8, 7], [8, 8], [25, 80]]) >>> clustering = DBSCAN(eps=3, min_samples=2).fit(X) >>> clustering.labels_ array([ 0, 0, 0, 1, 1, -1]) >>> clustering DBSCAN(eps=3, min_samples=2)
Пример см. в Демонстрация алгоритма кластеризации DBSCAN.
Для сравнения DBSCAN с другими алгоритмами кластеризации см. Сравнение различных алгоритмов кластеризации на игрушечных наборах данных
- fit(X, y=None, sample_weight=None)[источник]#
Выполнить кластеризацию DBSCAN по признакам или матрице расстояний.
- Параметры:
- X{array-like, sparse matrix} формы (n_samples, n_features) или (n_samples, n_samples)
Обучающие экземпляры для кластеризации, или расстояния между экземплярами, если
metric='precomputed'. Если предоставлена разреженная матрица, она будет преобразована в разреженнуюcsr_matrix.- yИгнорируется
Не используется, присутствует здесь для согласованности API по соглашению.
- sample_weightarray-like формы (n_samples,), по умолчанию=None
Вес каждой выборки, такой что выборка с весом не менее
min_samplesсам по себе является основным образцом; образец с отрицательным весом может препятствовать тому, чтобы его eps-сосед был основным. Обратите внимание, что веса абсолютные и по умолчанию равны 1.
- Возвращает:
- selfobject
Возвращает обученный экземпляр self.
- fit_predict(X, y=None, sample_weight=None)[источник]#
Вычисляет кластеры из матрицы данных или расстояний и предсказывает метки.
Этот метод обучает модель и возвращает метки кластеров за один шаг. Эквивалентно вызову fit(X).labels_.
- Параметры:
- X{array-like, sparse matrix} формы (n_samples, n_features) или (n_samples, n_samples)
Обучающие экземпляры для кластеризации, или расстояния между экземплярами, если
metric='precomputed'. Если предоставлена разреженная матрица, она будет преобразована в разреженнуюcsr_matrix.- yИгнорируется
Не используется, присутствует здесь для согласованности API по соглашению.
- sample_weightarray-like формы (n_samples,), по умолчанию=None
Вес каждой выборки, такой что выборка с весом не менее
min_samplesсам по себе является основным образцом; образец с отрицательным весом может препятствовать тому, чтобы его eps-сосед был основным. Обратите внимание, что веса абсолютные и по умолчанию равны 1.
- Возвращает:
- меткиndarray формы (n_samples,)
Метки кластеров. Зашумленные образцы получают метку -1. Неотрицательные целые числа указывают принадлежность к кластеру.
- 6332()[источник]#
Получить маршрутизацию метаданных этого объекта.
Пожалуйста, проверьте Руководство пользователя о том, как работает механизм маршрутизации.
- Возвращает:
- маршрутизацияMetadataRequest
A
MetadataRequestИнкапсуляция информации о маршрутизации.
- get_params(глубокий=True)[источник]#
Получить параметры для этого оценщика.
- Параметры:
- глубокийbool, по умолчанию=True
Если True, вернет параметры для этого оценщика и вложенных подобъектов, которые являются оценщиками.
- Возвращает:
- paramsdict
Имена параметров, сопоставленные с их значениями.
- set_fit_request(*, sample_weight: bool | None | str = '$UNCHANGED$') DBSCAN[источник]#
Настроить, следует ли запрашивать передачу метаданных в
fitметод.Обратите внимание, что этот метод актуален только тогда, когда этот оценщик используется как под-оценщик внутри мета-оценщик и маршрутизация метаданных включена с помощью
enable_metadata_routing=True(см.sklearn.set_config). Пожалуйста, проверьте Руководство пользователя о том, как работает механизм маршрутизации.Варианты для каждого параметра:
True: запрашиваются метаданные и передаютсяfitесли предоставлено. Запрос игнорируется, если метаданные не предоставлены.False: метаданные не запрашиваются, и мета-оценщик не передаст их вfit.None: метаданные не запрашиваются, и мета-оценщик выдаст ошибку, если пользователь предоставит их.str: метаданные должны передаваться мета-оценщику с этим заданным псевдонимом вместо исходного имени.
По умолчанию (
sklearn.utils.metadata_routing.UNCHANGED) сохраняет существующий запрос. Это позволяет изменять запрос для некоторых параметров, но не для других.Добавлено в версии 1.3.
- Параметры:
- sample_weightstr, True, False или None, по умолчанию=sklearn.utils.metadata_routing.UNCHANGED
Маршрутизация метаданных для
sample_weightпараметр вfit.
- Возвращает:
- selfobject
Обновленный объект.
- set_params(**params)[источник]#
Установить параметры этого оценщика.
Метод работает как на простых оценщиках, так и на вложенных объектах (таких как
Pipeline). Последние имеют параметры вида__ - Параметры:
- **paramsdict
Параметры оценщика.
- Возвращает:
- selfэкземпляр estimator
Экземпляр оценщика.
Примеры галереи#
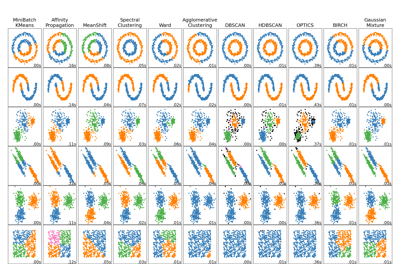
Сравнение различных алгоритмов кластеризации на игрушечных наборах данных