HDBSCAN#
- класс sklearn.cluster.HDBSCAN(min_cluster_size=5, min_samples=None, cluster_selection_epsilon=0.0, max_cluster_size=None, метрика='euclidean', metric_params=None, alpha=1.0, алгоритм='auto', leaf_size=40, n_jobs=None, cluster_selection_method='eom', allow_single_cluster=False, store_centers=None, copy='warn')[источник]#
Кластеризация данных с использованием иерархической кластеризации на основе плотности.
HDBSCAN - Иерархическая кластеризация на основе плотности пространственных приложений с шумом. Выполняет
DBSCANпо изменяющимся значениям epsilon и интегрирует результат, чтобы найти кластеризацию, дающую наилучшую стабильность по epsilon. Это позволяет HDBSCAN находить кластеры с разной плотностью (в отличие отDBSCAN), и быть более устойчивыми к выбору параметров. Подробнее в Руководство пользователя.Добавлено в версии 1.3.
- Параметры:
- min_cluster_sizeint, по умолчанию=5
Минимальное количество образцов в группе, чтобы эта группа считалась кластером; группировки меньшего размера будут оставлены как шум.
- min_samplesint, default=None
Параметр
kиспользуется для вычисления расстояния между точкойx_pи его k-й ближайший сосед. КогдаNone, по умолчаниюmin_cluster_size.- cluster_selection_epsilonfloat, по умолчанию=0.0
Порог расстояния. Кластеры ниже этого значения будут объединены. См. [5] для получения дополнительной информации.
- max_cluster_sizeint, default=None
Ограничение на размер кластеров, возвращаемых
"eom"алгоритм выбора кластеров. Нет ограничения, когдаmax_cluster_size=None. Не имеет эффекта, еслиcluster_selection_method="leaf".- метрикаstr или callable, по умолчанию='euclidean'
Метрика для вычисления расстояния между экземплярами в массиве признаков.
Если metric является строкой или вызываемым объектом, он должен быть одним из вариантов, разрешенных
pairwise_distancesдля своего параметра metric.Если метрика "precomputed", X считается матрицей расстояний и должна быть квадратной.
- metric_paramsdict, по умолчанию=None
Аргументы, передаваемые метрике расстояния.
- alphafloat, по умолчанию=1.0
Параметр масштабирования расстояния, используемый в устойчивой одиночной связи. См. [3] для получения дополнительной информации.
- алгоритм{“auto”, “brute”, “kd_tree”, “ball_tree”}, default=”auto”
Какой именно алгоритм использовать для вычисления основных расстояний; По умолчанию это установлено в
"auto"который пытается использоватьKDTreeдерево, если возможно, в противном случае используетсяBallTreeдерево. Оба"kd_tree"и"ball_tree"алгоритмы используютNearestNeighborsоценщик.Если
Xпереданный во времяfitявляется разреженным илиmetricнедействителен для обоихKDTreeиBallTree, тогда он разрешается использовать"brute"алгоритма.- leaf_sizeint, по умолчанию=40
Размер листа для деревьев, ответственных за быстрые запросы ближайших соседей, когда используются KDTree или BallTree как алгоритмы core-distance. Большой размер набора данных и маленький
leaf_sizeможет вызвать чрезмерное использование памяти. Если у вас заканчивается память, рассмотрите увеличениеleaf_sizeпараметр. Игнорируется дляalgorithm="brute".- n_jobsint, default=None
Количество задач для параллельного вычисления расстояний.
Noneозначает 1, если только не вjoblib.parallel_backendконтекст.-1означает использование всех процессоров. См. Глоссарий для получения дополнительной информации.- cluster_selection_method{“eom”, “leaf”}, по умолчанию “eom”
Метод, используемый для выбора кластеров из сжатого дерева. Стандартный подход для HDBSCAN* — использовать избыток массы (
"eom") алгоритм для поиска наиболее устойчивых кластеров. В качестве альтернативы вы можете выбрать кластеры на листьях дерева — это обеспечивает наиболее детализированные и однородные кластеры.- allow_single_clusterbool, по умолчанию=False
По умолчанию HDBSCAN* не будет создавать один кластер, установка этого в True переопределит это и позволит результаты с одним кластером в случае, если вы считаете это допустимым результатом для вашего набора данных.
- store_centersstr, default=None
Какие, если есть, центры кластеров вычислять и сохранять. Варианты:
Noneкоторый не вычисляет и не хранит центры."centroid"который вычисляет центр, взяв взвешенное среднее их позиций. Обратите внимание, что алгоритм использует евклидову метрику и не гарантирует, что результат будет наблюдаемой точкой данных."medoid"который вычисляет центр, беря точку в подогнанных данных, которая минимизирует расстояние до всех других точек в кластере. Это медленнее, чем "центроид", поскольку требует вычисления дополнительных попарных расстояний между точками одного кластера, но гарантирует, что выходные данные являются наблюдаемой точкой данных. Медиоид также хорошо определён для произвольных метрик и не зависит от евклидовой метрики."both"который вычисляет и сохраняет обе формы центров.
- copybool, по умолчанию=False
Если
copy=Trueтогда в любое время, когда будут сделаны изменения на месте, которые перезапишут данные, переданные в fit, сначала будет сделана копия, гарантируя, что исходные данные останутся неизменными. В настоящее время это применяется только когдаmetric="precomputed", при передаче плотного массива или разреженной матрицы CSR и когдаalgorithm="brute".Изменено в версии 1.10: Значение по умолчанию для
copyизменится сFalsetoTrueв версии 1.10.
- Атрибуты:
- labels_ndarray формы (n_samples,)
Метки кластеров для каждой точки в наборе данных, переданном в fit. Выбросы помечены следующим образом:
Зашумленные выборки получают метку -1.
Образцы с бесконечными элементами (+/- np.inf) получают метку -2.
Образцам с отсутствующими данными присваивается метка -3, даже если они также содержат бесконечные элементы.
- probabilities_ndarray формы (n_samples,)
Степень, с которой каждый образец является членом своего назначенного кластера.
Кластеризованные выборки имеют вероятности, пропорциональные степени их устойчивости как части кластера.
Зашумленные выборки имеют вероятность ноль.
Выборки с бесконечными элементами (+/- np.inf) имеют вероятность 0.
Образцы с отсутствующими данными имеют вероятность
np.nan.
- n_features_in_int
Количество признаков, замеченных во время fit.
- feature_names_in_ndarray формы (
n_features_in_,) Имена признаков, наблюдаемых во время fit. Определено только когда
Xимеет имена признаков, которые все являются строками.- centroids_ndarray формы (n_clusters, n_features)
Коллекция, содержащая центроид каждого кластера, вычисленный с использованием стандартной евклидовой метрики. Центроиды могут находиться "вне" своих соответствующих кластеров, если сами кластеры невыпуклые.
Обратите внимание, что
n_clustersучитывает только невыбросные кластеры. То есть,-1, -2, -3Метки для кластеров выбросов исключены.- medoids_ndarray формы (n_clusters, n_features)
Коллекция, содержащая медиоиды каждого кластера, вычисленные с использованием метрики, переданной в
metricпараметра. Медиоиды - это точки в исходном кластере, которые минимизируют среднее расстояние до всех других точек в этом кластере при выбранной метрике. Их можно рассматривать как результат проекцииmetric-based центроид обратно на кластер.Обратите внимание, что
n_clustersучитывает только невыбросные кластеры. То есть,-1, -2, -3Метки для кластеров выбросов исключены.
Смотрите также
Примечания
The
min_samplesпараметр включает саму точку, тогда как реализация в scikit-learn-contrib/hdbscan не делает. Чтобы получить одинаковые результаты в обеих версиях, значениеmin_samplesздесь должно быть на 1 больше значения, использованного в scikit-learn-contrib/hdbscan.Ссылки
Примеры
>>> import numpy as np >>> from sklearn.cluster import HDBSCAN >>> from sklearn.datasets import load_digits >>> X, _ = load_digits(return_X_y=True) >>> hdb = HDBSCAN(copy=True, min_cluster_size=20) >>> hdb.fit(X) HDBSCAN(copy=True, min_cluster_size=20) >>> hdb.labels_.shape == (X.shape[0],) True >>> np.unique(hdb.labels_).tolist() [-1, 0, 1, 2, 3, 4, 5, 6, 7]
- dbscan_clustering(cut_distance, min_cluster_size=5)[источник]#
Возвращает кластеризацию, полученную методом DBSCAN без граничных точек.
Возвращает кластеризацию, которая была бы эквивалентна запуску DBSCAN* для определенного cut_distance (или epsilon). DBSCAN* можно рассматривать как DBSCAN без граничных точек. Таким образом, эти результаты могут немного отличаться от
cluster.DBSCANиз-за различий в реализации для неосновных точек.Это также можно рассматривать как плоскую кластеризацию, полученную из разреза постоянной высоты через дерево одиночной связи.
Это представляет результат выбора порогового значения для устойчивой кластеризации с одиночной связью. The
min_cluster_sizeпозволяет плоской кластеризации объявлять точки шума (и кластеры меньшеmin_cluster_size).- Параметры:
- cut_distancefloat
Значение отсечки взаимной достижимости расстояния, используемое для генерации плоской кластеризации.
- min_cluster_sizeint, по умолчанию=5
Кластеры меньше этого значения будут называться 'шумом' и останутся некластеризованными в итоговой плоской кластеризации.
- Возвращает:
- меткиndarray формы (n_samples,)
Массив меток кластеров, по одной на точку данных. Выбросы помечаются следующим образом:
Зашумленные выборки получают метку -1.
Образцы с бесконечными элементами (+/- np.inf) получают метку -2.
Образцам с отсутствующими данными присваивается метка -3, даже если они также содержат бесконечные элементы.
- fit(X, y=None)[источник]#
Найти кластеры на основе иерархической кластеризации по плотности.
- Параметры:
- X{array-like, sparse matrix} формы (n_samples, n_features) или ndarray формы (n_samples, n_samples)
Массив признаков или массив расстояний между образцами, если
metric='precomputed'.- yNone
Игнорируется.
- Возвращает:
- selfobject
Возвращает self.
- fit_predict(X, y=None)[источник]#
Кластеризовать X и вернуть соответствующие метки кластеров.
- Параметры:
- X{array-like, sparse matrix} формы (n_samples, n_features) или ndarray формы (n_samples, n_samples)
Массив признаков или массив расстояний между образцами, если
metric='precomputed'.- yNone
Игнорируется.
- Возвращает:
- yndarray формы (n_samples,)
Метки кластеров.
- 6332()[источник]#
Получить маршрутизацию метаданных этого объекта.
Пожалуйста, проверьте Руководство пользователя о том, как работает механизм маршрутизации.
- Возвращает:
- маршрутизацияMetadataRequest
A
MetadataRequestИнкапсуляция информации о маршрутизации.
- get_params(глубокий=True)[источник]#
Получить параметры для этого оценщика.
- Параметры:
- глубокийbool, по умолчанию=True
Если True, вернет параметры для этого оценщика и вложенных подобъектов, которые являются оценщиками.
- Возвращает:
- paramsdict
Имена параметров, сопоставленные с их значениями.
- set_params(**params)[источник]#
Установить параметры этого оценщика.
Метод работает как на простых оценщиках, так и на вложенных объектах (таких как
Pipeline). Последние имеют параметры вида__ - Параметры:
- **paramsdict
Параметры оценщика.
- Возвращает:
- selfэкземпляр estimator
Экземпляр оценщика.
Примеры галереи#
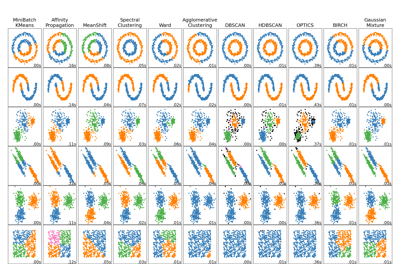
Сравнение различных алгоритмов кластеризации на игрушечных наборах данных