Примечание
Перейти в конец чтобы скачать полный пример кода или запустить этот пример в браузере через JupyterLite или Binder.
Важность перестановок с мультиколлинеарными или коррелированными признаками#
В этом примере мы вычисляем
permutation_importance признаков к обученной
RandomForestClassifier используя
Набор данных о раке молочной железы в Висконсине (диагностический). Модель может легко достичь точности около 97% на тестовом наборе данных. Поскольку этот набор данных содержит мультиколлинеарные признаки, перестановочная важность показывает, что ни один из признаков не является важным, что противоречит высокой тестовой точности.
Мы демонстрируем возможный подход к обработке мультиколлинеарности, который состоит из иерархической кластеризации по корреляциям Спирмена между признаками, выбора порога и сохранения одного признака из каждого кластера.
Примечание
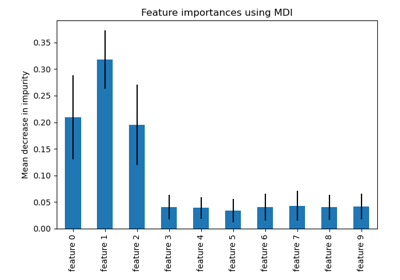
Смотрите также Важность перестановок против важности признаков случайного леса (MDI)
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Важность признаков случайного леса на данных о раке молочной железы#
Сначала определим функцию для упрощения построения графиков:
import matplotlib
from sklearn.inspection import permutation_importance
from sklearn.utils.fixes import parse_version
def plot_permutation_importance(clf, X, y, ax):
result = permutation_importance(clf, X, y, n_repeats=10, random_state=42, n_jobs=2)
perm_sorted_idx = result.importances_mean.argsort()
# `labels` argument in boxplot is deprecated in matplotlib 3.9 and has been
# renamed to `tick_labels`. The following code handles this, but as a
# scikit-learn user you probably can write simpler code by using `labels=...`
# (matplotlib < 3.9) or `tick_labels=...` (matplotlib >= 3.9).
tick_labels_parameter_name = (
"tick_labels"
if parse_version(matplotlib.__version__) >= parse_version("3.9")
else "labels"
)
tick_labels_dict = {tick_labels_parameter_name: X.columns[perm_sorted_idx]}
ax.boxplot(result.importances[perm_sorted_idx].T, vert=False, **tick_labels_dict)
ax.axvline(x=0, color="k", linestyle="--")
return ax
Затем мы обучаем RandomForestClassifier на
Набор данных о раке молочной железы в Висконсине (диагностический) и оценить его точность на тестовом наборе:
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
X, y = load_breast_cancer(return_X_y=True, as_frame=True)
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
clf = RandomForestClassifier(n_estimators=100, random_state=42)
clf.fit(X_train, y_train)
print(f"Baseline accuracy on test data: {clf.score(X_test, y_test):.2}")
Baseline accuracy on test data: 0.97
Далее мы строим важность признаков на основе дерева и важность перестановок. Важность перестановок рассчитывается на обучающем наборе, чтобы показать, насколько модель полагается на каждый признак во время обучения.
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
mdi_importances = pd.Series(clf.feature_importances_, index=X_train.columns)
tree_importance_sorted_idx = np.argsort(clf.feature_importances_)
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(12, 8))
mdi_importances.sort_values().plot.barh(ax=ax1)
ax1.set_xlabel("Gini importance")
plot_permutation_importance(clf, X_train, y_train, ax2)
ax2.set_xlabel("Decrease in accuracy score")
fig.suptitle(
"Impurity-based vs. permutation importances on multicollinear features (train set)"
)
_ = fig.tight_layout()
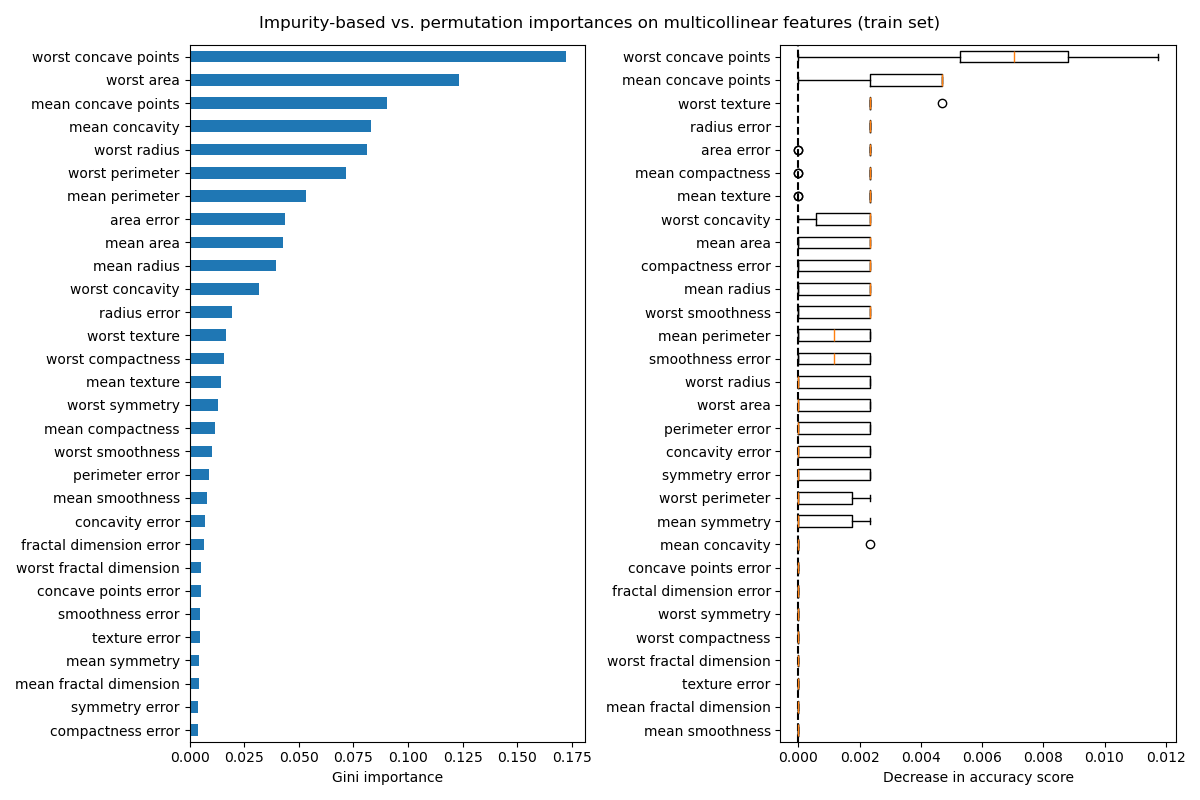
График слева показывает важность Джини модели. Поскольку
реализация scikit-learn
RandomForestClassifier использует случайные подмножества
\(\sqrt{n_\text{features}}\) признаков на каждом разделении, он способен размыть
доминирование любого одного коррелированного признака. В результате индивидуальная
важность признаков может распределяться более равномерно среди коррелированных
признаков. Поскольку признаки имеют большую мощность, а классификатор
не переобучен, мы можем относительно доверять этим значениям.
На графике справа важность перестановки показывает, что перестановка признака снижает точность не более чем 0.012, что может указывать на то, что ни один из признаков не важен. Это противоречит высокой тестовой точности, вычисленной как базовый уровень: некоторые признаки должны быть важными.
Аналогично, изменение оценки точности, вычисленной на тестовом наборе, по-видимому, обусловлено случайностью:
fig, ax = plt.subplots(figsize=(7, 6))
plot_permutation_importance(clf, X_test, y_test, ax)
ax.set_title("Permutation Importances on multicollinear features\n(test set)")
ax.set_xlabel("Decrease in accuracy score")
_ = ax.figure.tight_layout()
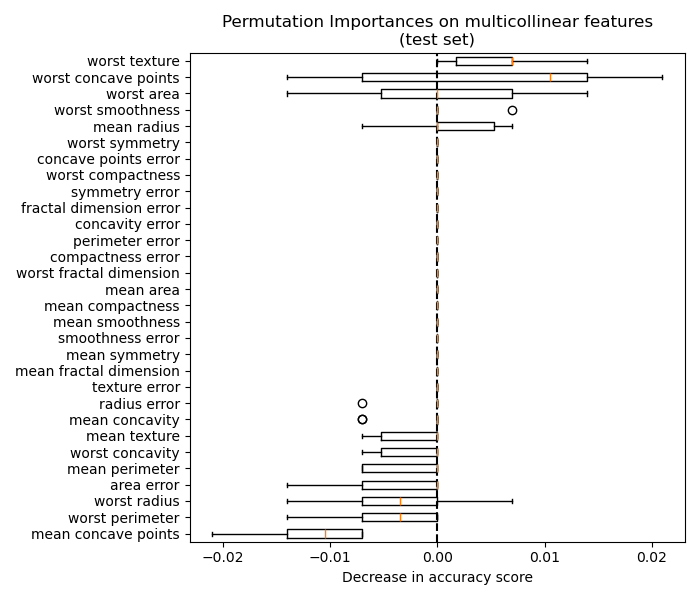
Тем не менее, можно все еще вычислить значимую важность перестановки в присутствии коррелированных признаков, как показано в следующем разделе.
Обработка мультиколлинеарных признаков#
Когда признаки коллинеарны, перестановка одного признака мало влияет на производительность модели, потому что она может получить ту же информацию из коррелированного признака. Обратите внимание, что это не относится ко всем прогностическим моделям и зависит от их базовой реализации.
Один из способов обработки мультиколлинеарных признаков — выполнение иерархической кластеризации на корреляциях Спирмена, выбор порога и сохранение одного признака из каждого кластера. Сначала мы строим тепловую карту коррелированных признаков:
from scipy.cluster import hierarchy
from scipy.spatial.distance import squareform
from scipy.stats import spearmanr
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(12, 8))
corr = spearmanr(X).correlation
# Ensure the correlation matrix is symmetric
corr = (corr + corr.T) / 2
np.fill_diagonal(corr, 1)
# We convert the correlation matrix to a distance matrix before performing
# hierarchical clustering using Ward's linkage.
distance_matrix = 1 - np.abs(corr)
dist_linkage = hierarchy.ward(squareform(distance_matrix))
dendro = hierarchy.dendrogram(
dist_linkage, labels=X.columns.to_list(), ax=ax1, leaf_rotation=90
)
dendro_idx = np.arange(0, len(dendro["ivl"]))
ax2.imshow(corr[dendro["leaves"], :][:, dendro["leaves"]])
ax2.set_xticks(dendro_idx)
ax2.set_yticks(dendro_idx)
ax2.set_xticklabels(dendro["ivl"], rotation="vertical")
ax2.set_yticklabels(dendro["ivl"])
_ = fig.tight_layout()
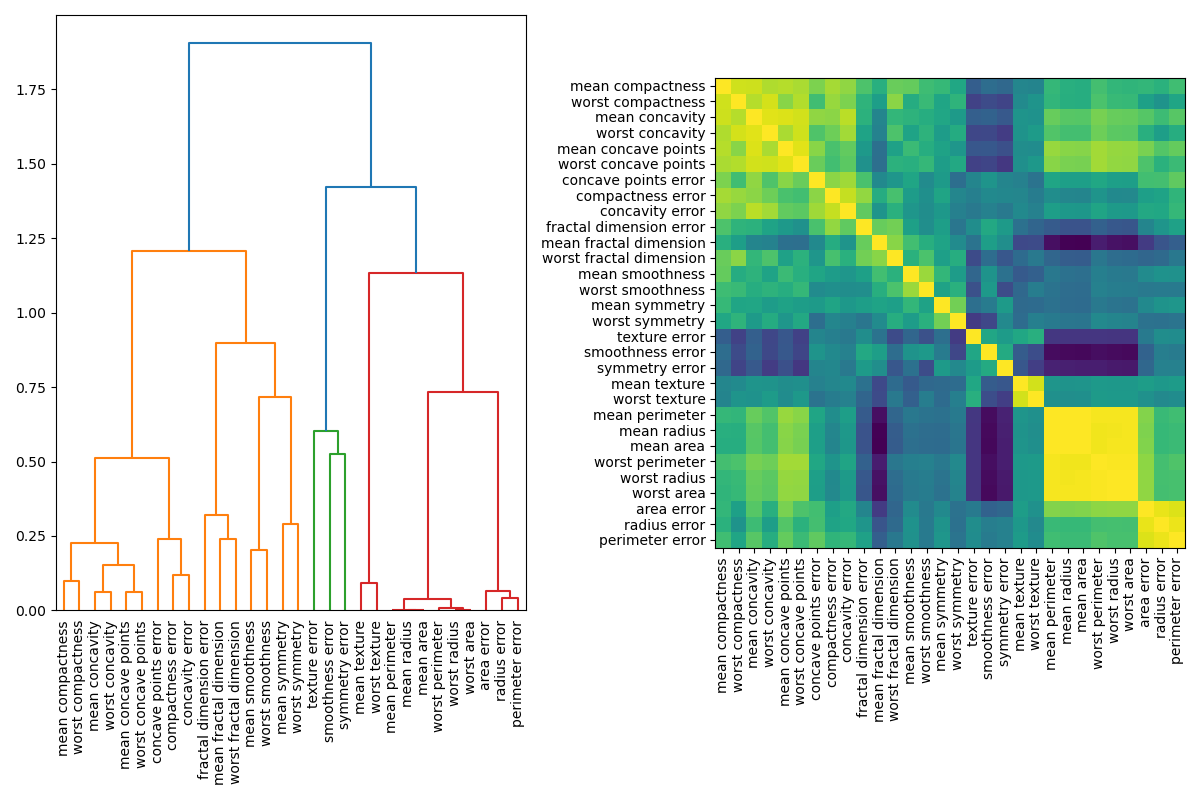
Далее мы вручную выбираем порог путем визуального осмотра дендрограммы, чтобы сгруппировать наши признаки в кластеры и выбрать по одному признаку из каждого кластера для сохранения, выбираем эти признаки из нашего набора данных и обучаем новый случайный лес. Тестовая точность нового случайного леса почти не изменилась по сравнению со случайным лесом, обученным на полном наборе данных.
from collections import defaultdict
cluster_ids = hierarchy.fcluster(dist_linkage, 1, criterion="distance")
cluster_id_to_feature_ids = defaultdict(list)
for idx, cluster_id in enumerate(cluster_ids):
cluster_id_to_feature_ids[cluster_id].append(idx)
selected_features = [v[0] for v in cluster_id_to_feature_ids.values()]
selected_features_names = X.columns[selected_features]
X_train_sel = X_train[selected_features_names]
X_test_sel = X_test[selected_features_names]
clf_sel = RandomForestClassifier(n_estimators=100, random_state=42)
clf_sel.fit(X_train_sel, y_train)
print(
"Baseline accuracy on test data with features removed:"
f" {clf_sel.score(X_test_sel, y_test):.2}"
)
Baseline accuracy on test data with features removed: 0.97
Наконец, мы можем исследовать важность перестановок выбранного подмножества признаков:
fig, ax = plt.subplots(figsize=(7, 6))
plot_permutation_importance(clf_sel, X_test_sel, y_test, ax)
ax.set_title("Permutation Importances on selected subset of features\n(test set)")
ax.set_xlabel("Decrease in accuracy score")
ax.figure.tight_layout()
plt.show()
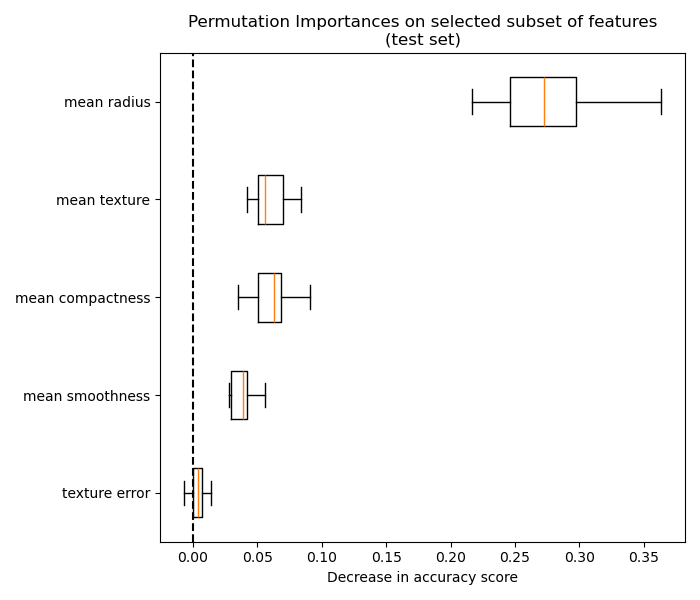
Общее время выполнения скрипта: (0 минут 8.771 секунд)
Связанные примеры
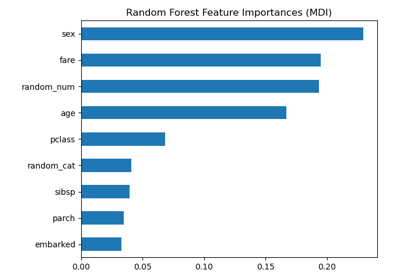
Важность перестановок против важности признаков случайного леса (MDI)