Примечание
Перейти в конец чтобы скачать полный пример кода или запустить этот пример в браузере через JupyterLite или Binder.
Методы обучения многообразий на разрезанной сфере#
Применение различных Обучение многообразию методов на сферическом наборе данных. Здесь можно увидеть использование снижения размерности для получения интуитивного понимания методов обучения многообразий. Что касается набора данных, полюса вырезаны из сферы, а также тонкий срез вдоль её стороны. Это позволяет методам обучения многообразий 'раскрыть' его при проекции на два измерения.
Для аналогичного примера, где методы применяются к набору данных S-кривой, см. Сравнение методов обучения многообразий.
Обратите внимание, что цель MDS заключается в нахождении низкоразмерного представления данных (здесь 2D), в котором расстояния хорошо соответствуют расстояниям в исходном высокоразмерном пространстве, в отличие от других алгоритмов обучения многообразиям, он не стремится к изотропному представлению данных в низкоразмерном пространстве. Здесь задача многообразия довольно хорошо соответствует задаче представления плоской карты Земли, как с картографическая проекция.
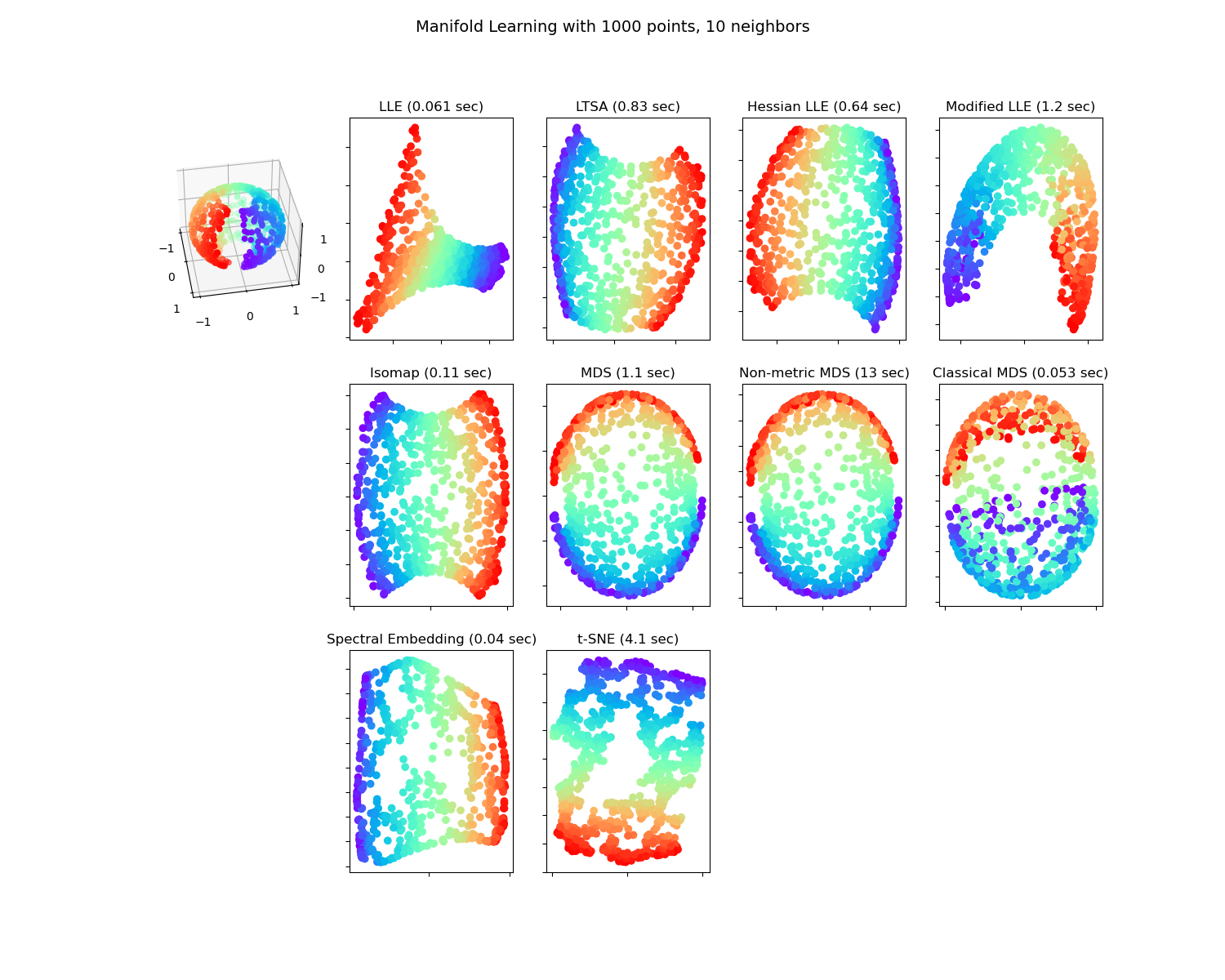
standard: 0.061 sec
ltsa: 0.83 sec
hessian: 0.64 sec
modified: 1.2 sec
ISO: 0.11 sec
MDS: 1.1 sec
Non-metric MDS: 13 sec
Classical MDS: 0.053 sec
Spectral Embedding: 0.04 sec
t-SNE: 4.1 sec
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
from time import time
import matplotlib.pyplot as plt
# Unused but required import for doing 3d projections with matplotlib < 3.2
import mpl_toolkits.mplot3d # noqa: F401
import numpy as np
from matplotlib.ticker import NullFormatter
from sklearn import manifold
from sklearn.utils import check_random_state
# Variables for manifold learning.
n_neighbors = 10
n_samples = 1000
# Create our sphere.
random_state = check_random_state(0)
p = random_state.rand(n_samples) * (2 * np.pi - 0.55)
t = random_state.rand(n_samples) * np.pi
# Sever the poles from the sphere.
indices = (t < (np.pi - (np.pi / 8))) & (t > (np.pi / 8))
colors = p[indices]
x, y, z = (
np.sin(t[indices]) * np.cos(p[indices]),
np.sin(t[indices]) * np.sin(p[indices]),
np.cos(t[indices]),
)
# Plot our dataset.
fig = plt.figure(figsize=(15, 12))
plt.suptitle(
"Manifold Learning with %i points, %i neighbors" % (1000, n_neighbors), fontsize=14
)
ax = fig.add_subplot(351, projection="3d")
ax.scatter(x, y, z, c=p[indices], cmap=plt.cm.rainbow)
ax.view_init(40, -10)
sphere_data = np.array([x, y, z]).T
# Perform Locally Linear Embedding Manifold learning
methods = ["standard", "ltsa", "hessian", "modified"]
labels = ["LLE", "LTSA", "Hessian LLE", "Modified LLE"]
for i, method in enumerate(methods):
t0 = time()
trans_data = (
manifold.LocallyLinearEmbedding(
n_neighbors=n_neighbors, n_components=2, method=method, random_state=42
)
.fit_transform(sphere_data)
.T
)
t1 = time()
print("%s: %.2g sec" % (methods[i], t1 - t0))
ax = fig.add_subplot(352 + i)
plt.scatter(trans_data[0], trans_data[1], c=colors, cmap=plt.cm.rainbow)
plt.title("%s (%.2g sec)" % (labels[i], t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter())
ax.yaxis.set_major_formatter(NullFormatter())
plt.axis("tight")
# Perform Isomap Manifold learning.
t0 = time()
trans_data = (
manifold.Isomap(n_neighbors=n_neighbors, n_components=2)
.fit_transform(sphere_data)
.T
)
t1 = time()
print("%s: %.2g sec" % ("ISO", t1 - t0))
ax = fig.add_subplot(357)
plt.scatter(trans_data[0], trans_data[1], c=colors, cmap=plt.cm.rainbow)
plt.title("%s (%.2g sec)" % ("Isomap", t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter())
ax.yaxis.set_major_formatter(NullFormatter())
plt.axis("tight")
# Perform Multi-dimensional scaling.
t0 = time()
mds = manifold.MDS(2, n_init=1, random_state=42, init="classical_mds")
trans_data = mds.fit_transform(sphere_data).T
t1 = time()
print("MDS: %.2g sec" % (t1 - t0))
ax = fig.add_subplot(358)
plt.scatter(trans_data[0], trans_data[1], c=colors, cmap=plt.cm.rainbow)
plt.title("MDS (%.2g sec)" % (t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter())
ax.yaxis.set_major_formatter(NullFormatter())
plt.axis("tight")
t0 = time()
mds = manifold.MDS(2, n_init=1, random_state=42, metric_mds=False, init="classical_mds")
trans_data = mds.fit_transform(sphere_data).T
t1 = time()
print("Non-metric MDS: %.2g sec" % (t1 - t0))
ax = fig.add_subplot(359)
plt.scatter(trans_data[0], trans_data[1], c=colors, cmap=plt.cm.rainbow)
plt.title("Non-metric MDS (%.2g sec)" % (t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter())
ax.yaxis.set_major_formatter(NullFormatter())
plt.axis("tight")
t0 = time()
mds = manifold.ClassicalMDS(2)
trans_data = mds.fit_transform(sphere_data).T
t1 = time()
print("Classical MDS: %.2g sec" % (t1 - t0))
ax = fig.add_subplot(3, 5, 10)
plt.scatter(trans_data[0], trans_data[1], c=colors, cmap=plt.cm.rainbow)
plt.title("Classical MDS (%.2g sec)" % (t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter())
ax.yaxis.set_major_formatter(NullFormatter())
plt.axis("tight")
# Perform Spectral Embedding.
t0 = time()
se = manifold.SpectralEmbedding(
n_components=2, n_neighbors=n_neighbors, random_state=42
)
trans_data = se.fit_transform(sphere_data).T
t1 = time()
print("Spectral Embedding: %.2g sec" % (t1 - t0))
ax = fig.add_subplot(3, 5, 12)
plt.scatter(trans_data[0], trans_data[1], c=colors, cmap=plt.cm.rainbow)
plt.title("Spectral Embedding (%.2g sec)" % (t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter())
ax.yaxis.set_major_formatter(NullFormatter())
plt.axis("tight")
# Perform t-distributed stochastic neighbor embedding.
t0 = time()
tsne = manifold.TSNE(n_components=2, random_state=0)
trans_data = tsne.fit_transform(sphere_data).T
t1 = time()
print("t-SNE: %.2g sec" % (t1 - t0))
ax = fig.add_subplot(3, 5, 13)
plt.scatter(trans_data[0], trans_data[1], c=colors, cmap=plt.cm.rainbow)
plt.title("t-SNE (%.2g sec)" % (t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter())
ax.yaxis.set_major_formatter(NullFormatter())
plt.axis("tight")
plt.show()
Общее время выполнения скрипта: (0 минут 21.806 секунд)
Связанные примеры
t-SNE: Влияние различных значений perplexity на форму

Обучение многообразию на рукописных цифрах: Locally Linear Embedding, Isomap…