Примечание
Перейти в конец чтобы скачать полный пример кода или запустить этот пример в браузере через JupyterLite или Binder.
Заполнение пропущенных значений перед построением оценщика#
Пропущенные значения могут быть заменены средним, медианой или наиболее частым значением с использованием базового SimpleImputer.
В этом примере мы исследуем различные методы импутации:
заполнение пропусков постоянным значением 0
заполнение средним значением каждого признака
k ближайших соседей импутация
итеративное заполнение пропусков
Во всех случаях для каждого признака мы добавляем новый признак, указывающий на пропущенные значения.
Мы будем использовать два набора данных: набор данных Diabetes, который состоит из 10 признаков, собранных у пациентов с диабетом, с целью прогнозирования прогрессирования заболевания, и набор данных California housing, для которого целью является медианная стоимость жилья для районов Калифорнии.
Поскольку ни один из этих наборов данных не имеет пропущенных значений, мы удалим некоторые значения, чтобы создать новые версии с искусственно пропущенными данными. Производительность
RandomForestRegressor на полном исходном наборе данных затем сравнивается с производительностью на изменённых наборах данных с искусственно пропущенными значениями, импутированными с использованием различных техник.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Загрузить данные и создать наборы пропущенных значений#
Сначала мы загружаем два набора данных. Набор данных Diabetes поставляется с scikit-learn. Он содержит 442 записи, каждая с 10 признаками. Набор данных California housing намного больше с 20640 записями и 8 признаками. Его нужно загрузить. Мы будем использовать только первые 300 записей для ускорения вычислений, но вы можете использовать весь набор данных.
import numpy as np
from sklearn.datasets import fetch_california_housing, load_diabetes
X_diabetes, y_diabetes = load_diabetes(return_X_y=True)
X_california, y_california = fetch_california_housing(return_X_y=True)
X_diabetes = X_diabetes[:300]
y_diabetes = y_diabetes[:300]
X_california = X_california[:300]
y_california = y_california[:300]
def add_missing_values(X_full, y_full, rng):
n_samples, n_features = X_full.shape
# Add missing values in 75% of the lines
missing_rate = 0.75
n_missing_samples = int(n_samples * missing_rate)
missing_samples = np.zeros(n_samples, dtype=bool)
missing_samples[:n_missing_samples] = True
rng.shuffle(missing_samples)
missing_features = rng.randint(0, n_features, n_missing_samples)
X_missing = X_full.copy()
X_missing[missing_samples, missing_features] = np.nan
y_missing = y_full.copy()
return X_missing, y_missing
rng = np.random.RandomState(42)
X_miss_diabetes, y_miss_diabetes = add_missing_values(X_diabetes, y_diabetes, rng)
X_miss_california, y_miss_california = add_missing_values(
X_california, y_california, rng
)
Заполнить пропущенные данные и оценить#
Теперь мы напишем функцию, которая будет оценивать результаты на по-разному импутированных данных, включая случай отсутствия импутации для полных данных. Мы будем использовать RandomForestRegressor для целевой
регрессии.
from sklearn.ensemble import RandomForestRegressor
# To use the experimental IterativeImputer, we need to explicitly ask for it:
from sklearn.experimental import enable_iterative_imputer # noqa: F401
from sklearn.impute import IterativeImputer, KNNImputer, SimpleImputer
from sklearn.model_selection import cross_val_score
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import RobustScaler
N_SPLITS = 4
def get_score(X, y, imputer=None):
regressor = RandomForestRegressor(random_state=0)
if imputer is not None:
estimator = make_pipeline(imputer, regressor)
else:
estimator = regressor
scores = cross_val_score(
estimator, X, y, scoring="neg_mean_squared_error", cv=N_SPLITS
)
return scores.mean(), scores.std()
x_labels = []
mses_diabetes = np.zeros(5)
stds_diabetes = np.zeros(5)
mses_california = np.zeros(5)
stds_california = np.zeros(5)
Оценить оценку#
Сначала мы хотим оценить оценку на исходных данных:
mses_diabetes[0], stds_diabetes[0] = get_score(X_diabetes, y_diabetes)
mses_california[0], stds_california[0] = get_score(X_california, y_california)
x_labels.append("Full Data")
Заменить пропущенные значения на 0#
Теперь мы оценим оценку на данных, где пропущенные значения заменены на 0:
imputer = SimpleImputer(strategy="constant", fill_value=0, add_indicator=True)
mses_diabetes[1], stds_diabetes[1] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[1], stds_california[1] = get_score(
X_miss_california, y_miss_california, imputer
)
x_labels.append("Zero Imputation")
Заполнить пропущенные значения средним#
imputer = SimpleImputer(strategy="mean", add_indicator=True)
mses_diabetes[2], stds_diabetes[2] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[2], stds_california[2] = get_score(
X_miss_california, y_miss_california, imputer
)
x_labels.append("Mean Imputation")
kNN-импутация пропущенных значений#
KNNImputer заполняет пропущенные значения с использованием взвешенного или невзвешенного среднего желаемого количества ближайших соседей. Если ваши признаки имеют сильно различающиеся масштабы (как в наборе данных California housing), рассмотрите их повторное масштабирование для потенциального улучшения производительности.
imputer = KNNImputer(add_indicator=True)
mses_diabetes[3], stds_diabetes[3] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[3], stds_california[3] = get_score(
X_miss_california, y_miss_california, make_pipeline(RobustScaler(), imputer)
)
x_labels.append("KNN Imputation")
Итеративная импутация пропущенных значений#
Другой вариант - это IterativeImputer. Это использует
циклическую регрессию, моделируя каждый признак с пропущенными значениями как
функцию других признаков, по очереди. Мы используем выбор по умолчанию
класса для модели регрессора (BayesianRidge) для предсказания отсутствующих значений признаков. Производительность предсказателя
может негативно сказаться из-за сильно различающихся масштабов признаков,
поэтому мы перемасштабируем признаки в наборе данных California housing.
imputer = IterativeImputer(add_indicator=True)
mses_diabetes[4], stds_diabetes[4] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[4], stds_california[4] = get_score(
X_miss_california, y_miss_california, make_pipeline(RobustScaler(), imputer)
)
x_labels.append("Iterative Imputation")
mses_diabetes = mses_diabetes * -1
mses_california = mses_california * -1
Построить график результатов#
Наконец, мы визуализируем оценку:
import matplotlib.pyplot as plt
n_bars = len(mses_diabetes)
xval = np.arange(n_bars)
colors = ["r", "g", "b", "orange", "black"]
# plot diabetes results
plt.figure(figsize=(12, 6))
ax1 = plt.subplot(121)
for j in xval:
ax1.barh(
j,
mses_diabetes[j],
xerr=stds_diabetes[j],
color=colors[j],
alpha=0.6,
align="center",
)
ax1.set_title("Imputation Techniques with Diabetes Data")
ax1.set_xlim(left=np.min(mses_diabetes) * 0.9, right=np.max(mses_diabetes) * 1.1)
ax1.set_yticks(xval)
ax1.set_xlabel("MSE")
ax1.invert_yaxis()
ax1.set_yticklabels(x_labels)
# plot california dataset results
ax2 = plt.subplot(122)
for j in xval:
ax2.barh(
j,
mses_california[j],
xerr=stds_california[j],
color=colors[j],
alpha=0.6,
align="center",
)
ax2.set_title("Imputation Techniques with California Data")
ax2.set_yticks(xval)
ax2.set_xlabel("MSE")
ax2.invert_yaxis()
ax2.set_yticklabels([""] * n_bars)
plt.show()
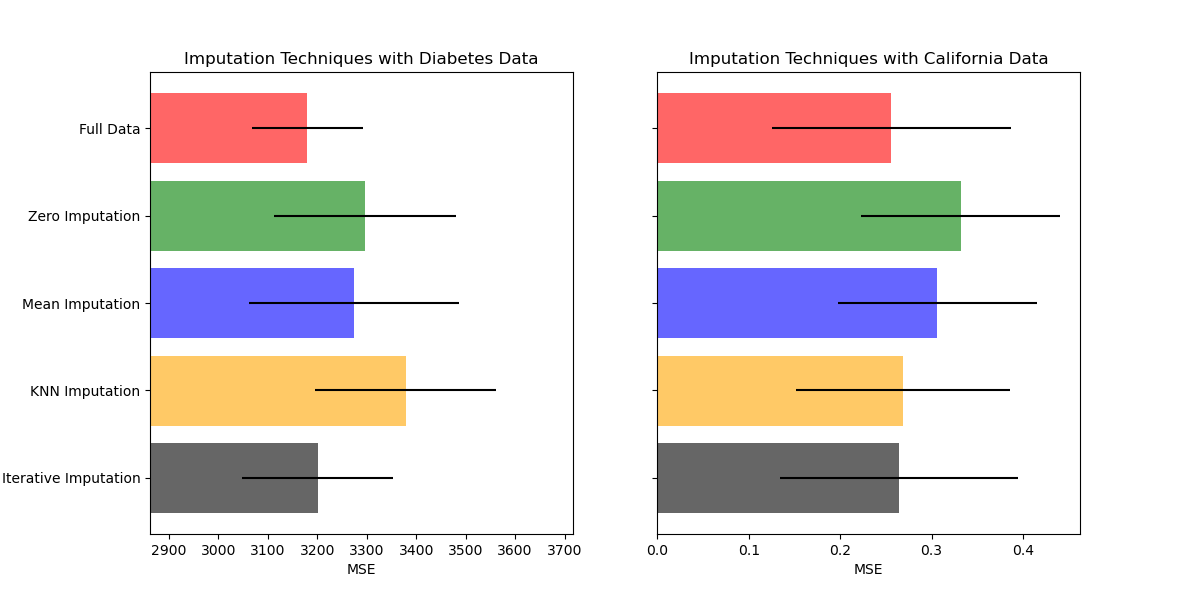
Вы также можете попробовать различные техники. Например, медиана является более устойчивой оценкой для данных с переменными большой величины, которые могут доминировать в результатах (также известными как 'длинный хвост').
Общее время выполнения скрипта: (0 минут 8.137 секунд)
Связанные примеры
Заполнение пропущенных значений с вариантами IterativeImputer
Анализ вариации байесовской гауссовой смеси с априорным типом концентрации