Примечание
Перейти в конец чтобы скачать полный пример кода или запустить этот пример в браузере через JupyterLite или Binder.
Масштабируемое обучение с полиномиальной аппроксимацией ядра#
Этот пример иллюстрирует использование PolynomialCountSketch для
эффективной генерации аппроксимаций пространства признаков полиномиального ядра.
Это используется для обучения линейных классификаторов, которые приближают точность
ядерных.
Мы используем набор данных Covtype [2], пытаясь воспроизвести эксперименты из
оригинальной статьи Tensor Sketch [1], т.е. алгоритм, реализованный в
PolynomialCountSketch.
Сначала мы вычисляем точность линейного классификатора на исходных
признаках. Затем мы обучаем линейные классификаторы на разном количестве
признаков (n_components) сгенерированный PolynomialCountSketch,
аппроксимируя точность ядерного классификатора масштабируемым способом.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Подготовка данных#
Загрузим набор данных Covtype, который содержит 581 012 образцов с 54 признаками каждый, распределённых среди 6 классов. Цель этого набора данных — предсказать тип лесного покрова только на основе картографических переменных (без данных дистанционного зондирования). После загрузки преобразуем его в задачу бинарной классификации, чтобы соответствовать версии набора данных на веб-странице LIBSVM. [2], который использовался в [1].
from sklearn.datasets import fetch_covtype
X, y = fetch_covtype(return_X_y=True)
y[y != 2] = 0
y[y == 2] = 1 # We will try to separate class 2 from the other 6 classes.
Разделение данных#
Здесь мы выбираем 5 000 выборок для обучения и 10 000 для тестирования. Чтобы фактически воспроизвести результаты в оригинальной статье Tensor Sketch, выберите 100 000 для обучения.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
X, y, train_size=5_000, test_size=10_000, random_state=42
)
Нормализация признаков#
Теперь масштабируем признаки до диапазона [0, 1], чтобы соответствовать формату набора данных на веб-странице LIBSVM, а затем нормализуем до единичной длины, как в оригинальной статье Tensor Sketch. [1].
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import MinMaxScaler, Normalizer
mm = make_pipeline(MinMaxScaler(), Normalizer())
X_train = mm.fit_transform(X_train)
X_test = mm.transform(X_test)
Установление базовой модели#
В качестве базового уровня обучите линейный SVM на исходных признаках и выведите точность. Мы также измеряем и сохраняем точность и время обучения для последующего построения графиков.
import time
from sklearn.svm import LinearSVC
results = {}
lsvm = LinearSVC()
start = time.time()
lsvm.fit(X_train, y_train)
lsvm_time = time.time() - start
lsvm_score = 100 * lsvm.score(X_test, y_test)
results["LSVM"] = {"time": lsvm_time, "score": lsvm_score}
print(f"Linear SVM score on raw features: {lsvm_score:.2f}%")
Linear SVM score on raw features: 75.62%
Создание модели аппроксимации ядра#
Затем мы обучаем линейные SVM на признаках, сгенерированных
PolynomialCountSketch с разными значениями для n_components,
показывая, что эти аппроксимации ядерных признаков улучшают точность
линейной классификации. В типичных сценариях применения, n_components
должно быть больше количества признаков во входном представлении, чтобы достичь улучшения по сравнению с линейной классификацией. Как правило, оптимальное значение оценки / стоимости времени выполнения обычно достигается примерно при n_components = 10 * n_features, хотя это
может зависеть от конкретного обрабатываемого набора данных. Обратите внимание, что, поскольку
исходные образцы имеют 54 признака, явное отображение признаков
полиномиального ядра четвертой степени будет иметь примерно 8,5 миллионов
признаков (точнее, 54^4). Благодаря PolynomialCountSketch, мы можем
сжать большую часть дискриминативной информации этого пространства признаков в
гораздо более компактное представление. Хотя мы проводим эксперимент только один раз
(n_runs = 1) в этом примере, на практике следует повторить эксперимент несколько раз, чтобы компенсировать стохастическую природу PolynomialCountSketch.
from sklearn.kernel_approximation import PolynomialCountSketch
n_runs = 1
N_COMPONENTS = [250, 500, 1000, 2000]
for n_components in N_COMPONENTS:
ps_lsvm_time = 0
ps_lsvm_score = 0
for _ in range(n_runs):
pipeline = make_pipeline(
PolynomialCountSketch(n_components=n_components, degree=4),
LinearSVC(),
)
start = time.time()
pipeline.fit(X_train, y_train)
ps_lsvm_time += time.time() - start
ps_lsvm_score += 100 * pipeline.score(X_test, y_test)
ps_lsvm_time /= n_runs
ps_lsvm_score /= n_runs
results[f"LSVM + PS({n_components})"] = {
"time": ps_lsvm_time,
"score": ps_lsvm_score,
}
print(
f"Linear SVM score on {n_components} PolynomialCountSketch "
f"features: {ps_lsvm_score:.2f}%"
)
Linear SVM score on 250 PolynomialCountSketch features: 76.55%
Linear SVM score on 500 PolynomialCountSketch features: 76.92%
Linear SVM score on 1000 PolynomialCountSketch features: 77.79%
Linear SVM score on 2000 PolynomialCountSketch features: 78.59%
Создание модели ядерного метода опорных векторов#
Обучите ядерный SVM, чтобы увидеть, насколько хорошо PolynomialCountSketch
аппроксимирует производительность ядра. Это, конечно, может занять некоторое время, так как класс SVC имеет относительно плохую масштабируемость. Вот почему аппроксиматоры ядра так полезны:
from sklearn.svm import SVC
ksvm = SVC(C=500.0, kernel="poly", degree=4, coef0=0, gamma=1.0)
start = time.time()
ksvm.fit(X_train, y_train)
ksvm_time = time.time() - start
ksvm_score = 100 * ksvm.score(X_test, y_test)
results["KSVM"] = {"time": ksvm_time, "score": ksvm_score}
print(f"Kernel-SVM score on raw features: {ksvm_score:.2f}%")
Kernel-SVM score on raw features: 79.78%
Сравнение результатов#
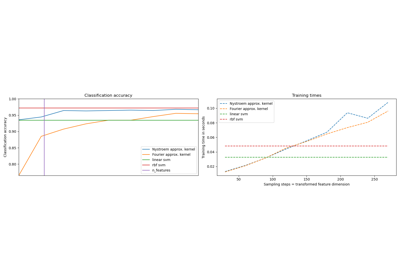
Наконец, построим график результатов различных методов в зависимости от времени их обучения. Как мы видим, ядерный SVM достигает более высокой точности, но время его обучения значительно больше и, что важнее всего, будет расти гораздо быстрее при увеличении количества обучающих выборок.
import matplotlib.pyplot as plt
fig, ax = plt.subplots(figsize=(7, 7))
ax.scatter(
[
results["LSVM"]["time"],
],
[
results["LSVM"]["score"],
],
label="Linear SVM",
c="green",
marker="^",
)
ax.scatter(
[
results["LSVM + PS(250)"]["time"],
],
[
results["LSVM + PS(250)"]["score"],
],
label="Linear SVM + PolynomialCountSketch",
c="blue",
)
for n_components in N_COMPONENTS:
ax.scatter(
[
results[f"LSVM + PS({n_components})"]["time"],
],
[
results[f"LSVM + PS({n_components})"]["score"],
],
c="blue",
)
ax.annotate(
f"n_comp.={n_components}",
(
results[f"LSVM + PS({n_components})"]["time"],
results[f"LSVM + PS({n_components})"]["score"],
),
xytext=(-30, 10),
textcoords="offset pixels",
)
ax.scatter(
[
results["KSVM"]["time"],
],
[
results["KSVM"]["score"],
],
label="Kernel SVM",
c="red",
marker="x",
)
ax.set_xlabel("Training time (s)")
ax.set_ylabel("Accuracy (%)")
ax.legend()
plt.show()

Ссылки#
Общее время выполнения скрипта: (0 минут 41.185 секунд)
Связанные примеры
Аппроксимация явного отображения признаков для RBF-ядер
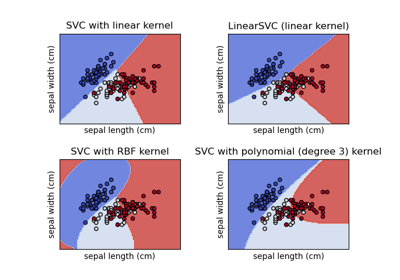
Построение различных классификаторов SVM на наборе данных iris