Примечание
Перейти в конец чтобы скачать полный пример кода или запустить этот пример в браузере через JupyterLite или Binder.
Оценка оценщиков обнаружения выбросов#
Этот пример сравнивает два алгоритма обнаружения выбросов, а именно
Локальный фактор выбросов (LOF) и Isolation Forest (IForest), на
реальных наборах данных, доступных в sklearn.datasets. Цель — показать,
что разные алгоритмы хорошо работают на разных наборах данных и сравнить их
скорость обучения и чувствительность к гиперпараметрам.
Алгоритмы обучаются (без меток) на всем наборе данных, который предполагается содержащим выбросы.
Кривые ROC вычисляются с использованием знания истинных меток и отображаются с помощью
RocCurveDisplay.Производительность оценивается с точки зрения ROC-AUC.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Предобработка набора данных и обучение модели#
Различные модели обнаружения выбросов требуют различной предобработки. При наличии категориальных переменных,
OrdinalEncoder часто является хорошей стратегией для моделей на основе деревьев, таких как IsolationForest, тогда как модели на основе соседей, такие как LocalOutlierFactor
могут быть затронуты упорядочиванием, индуцированным порядковым кодированием. Чтобы избежать индуцирования упорядочивания, следует использовать
OneHotEncoder.
Модели на основе соседей также могут требовать масштабирования числовых признаков (см., например, Влияние масштабирования на модели k-ближайших соседей). При наличии выбросов хорошим вариантом является использование RobustScaler.
from sklearn.compose import ColumnTransformer
from sklearn.ensemble import IsolationForest
from sklearn.neighbors import LocalOutlierFactor
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import (
OneHotEncoder,
OrdinalEncoder,
RobustScaler,
)
def make_estimator(name, categorical_columns=None, iforest_kw=None, lof_kw=None):
"""Create an outlier detection estimator based on its name."""
if name == "LOF":
outlier_detector = LocalOutlierFactor(**(lof_kw or {}))
if categorical_columns is None:
preprocessor = RobustScaler()
else:
preprocessor = ColumnTransformer(
transformers=[("categorical", OneHotEncoder(), categorical_columns)],
remainder=RobustScaler(),
)
else: # name == "IForest"
outlier_detector = IsolationForest(**(iforest_kw or {}))
if categorical_columns is None:
preprocessor = None
else:
ordinal_encoder = OrdinalEncoder(
handle_unknown="use_encoded_value", unknown_value=-1
)
preprocessor = ColumnTransformer(
transformers=[
("categorical", ordinal_encoder, categorical_columns),
],
remainder="passthrough",
)
return make_pipeline(preprocessor, outlier_detector)
Следующие fit_predict функция возвращает средний показатель аномалий для X.
from time import perf_counter
def fit_predict(estimator, X):
tic = perf_counter()
if estimator[-1].__class__.__name__ == "LocalOutlierFactor":
estimator.fit(X)
y_score = estimator[-1].negative_outlier_factor_
else: # "IsolationForest"
y_score = estimator.fit(X).decision_function(X)
toc = perf_counter()
print(f"Duration for {model_name}: {toc - tic:.2f} s")
return y_score
В остальной части примера мы обрабатываем один набор данных за раздел. После загрузки
данных цели изменяются, чтобы состоять из двух классов: 0, представляющий
внутренние точки, и 1, представляющий выбросы. Из-за вычислительных ограничений
документации scikit-learn, размер выборки некоторых наборов данных уменьшается с использованием
стратифицированного train_test_split.
Кроме того, мы устанавливаем n_neighbors для соответствия ожидаемому количеству аномалий
expected_n_anomalies = n_samples * expected_anomaly_fraction. Это хорошая
эвристика, пока доля выбросов не очень мала, причина
в том, что n_neighbors должно быть как минимум больше, чем количество образцов
в менее населенном кластере (см.
Обнаружение выбросов с помощью фактора локальных выбросов (LOF)).
KDDCup99 - SA dataset#
The Набор данных Kddcup 99 был сгенерирован с использованием закрытой сети и вручную внедренных атак. Набор данных SA является его подмножеством, полученным простым выбором всех нормальных данных и доли аномалий около 3%.
import numpy as np
from sklearn.datasets import fetch_kddcup99
from sklearn.model_selection import train_test_split
X, y = fetch_kddcup99(
subset="SA", percent10=True, random_state=42, return_X_y=True, as_frame=True
)
y = (y != b"normal.").astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.1, stratify=y, random_state=42)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
10065 datapoints with 338 anomalies (3.36%)
Набор данных SA содержит 41 признак, из которых 3 являются категориальными: “protocol_type”, “service” и “flag”.
y_true = {}
y_score = {"LOF": {}, "IForest": {}}
model_names = ["LOF", "IForest"]
cat_columns = ["protocol_type", "service", "flag"]
y_true["KDDCup99 - SA"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_score[model_name]["KDDCup99 - SA"] = fit_predict(model, X)
Duration for LOF: 1.75 s
Duration for IForest: 0.27 s
Набор данных Forest covertypes#
The Forest covertypes является многоклассовым набором данных, где целевая переменная — доминирующий вид дерева на данном участке леса. Он содержит 54 признака, некоторые из которых («Wilderness_Area» и «Soil_Type») уже бинарно закодированы. Хотя изначально предназначен для задачи классификации, можно рассматривать выбросы как образцы с меткой 2, а аномалии — с меткой 4.
from sklearn.datasets import fetch_covtype
X, y = fetch_covtype(return_X_y=True, as_frame=True)
s = (y == 2) + (y == 4)
X = X.loc[s]
y = y.loc[s]
y = (y != 2).astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.05, stratify=y, random_state=42)
X_forestcover = X # save X for later use
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
14302 datapoints with 137 anomalies (0.96%)
y_true["forestcover"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_score[model_name]["forestcover"] = fit_predict(model, X)
Duration for LOF: 1.72 s
Duration for IForest: 0.21 s
Набор данных Ames Housing#
The Набор данных о жилье в Эймсе изначально является набором данных регрессии, где целевой переменной являются цены продажи домов в Эймсе, Айова. Здесь мы преобразуем его в задачу обнаружения выбросов, рассматривая дома с ценой выше 70 долларов США за кв. фут. Чтобы упростить задачу, мы исключаем промежуточные цены между 40 и 70 долларами США за кв. фут.
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_openml
X, y = fetch_openml(name="ames_housing", version=1, return_X_y=True, as_frame=True)
y = y.div(X["Lot_Area"])
# None values in pandas 1.5.1 were mapped to np.nan in pandas 2.0.1
X["Misc_Feature"] = X["Misc_Feature"].cat.add_categories("NoInfo").fillna("NoInfo")
X["Mas_Vnr_Type"] = X["Mas_Vnr_Type"].cat.add_categories("NoInfo").fillna("NoInfo")
X.drop(columns="Lot_Area", inplace=True)
mask = (y < 40) | (y > 70)
X = X.loc[mask]
y = y.loc[mask]
y.hist(bins=20, edgecolor="black")
plt.xlabel("House price in USD/sqft")
_ = plt.title("Distribution of house prices in Ames")
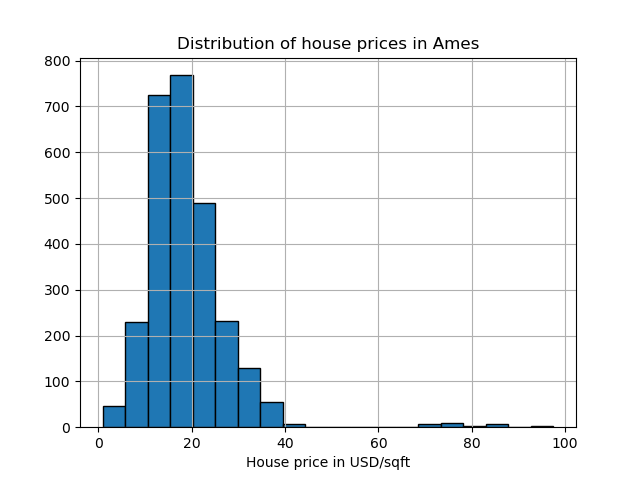
y = (y > 70).astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2714 datapoints with 30 anomalies (1.11%)
Набор данных содержит 46 категориальных признаков. В этом случае проще использовать
make_column_selector чтобы найти их вместо передачи
списка, созданного вручную.
from sklearn.compose import make_column_selector as selector
categorical_columns_selector = selector(dtype_include="category")
cat_columns = categorical_columns_selector(X)
y_true["ames_housing"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_score[model_name]["ames_housing"] = fit_predict(model, X)
Duration for LOF: 0.43 s
Duration for IForest: 0.21 s
Набор данных кардиотокографии#
The Набор данных кардиотокографии теперь поддерживает монотонные ограничения, полезные, когда признаки предположительно оказывают положительное/отрицательное влияние на целевую переменную.
X, y = fetch_openml(name="cardiotocography", version=1, return_X_y=True, as_frame=False)
X_cardiotocography = X # save X for later use
s = y == "3"
y = s.astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2126 datapoints with 53 anomalies (2.49%)
y_true["cardiotocography"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_score[model_name]["cardiotocography"] = fit_predict(model, X)
Duration for LOF: 0.05 s
Duration for IForest: 0.13 s
Построить и интерпретировать результаты#
Производительность алгоритма связана с тем, насколько хороша истинная положительная ставка (TPR) при низком значении ложной положительной ставки (FPR). Лучшие алгоритмы имеют кривую в верхнем левом углу графика и площадь под кривой (AUC), близкую к 1. Пунктирная диагональная линия представляет случайную классификацию выбросов и внутренних точек.
import math
from sklearn.metrics import RocCurveDisplay
cols = 2
pos_label = 0 # mean 0 belongs to positive class
datasets_names = y_true.keys()
rows = math.ceil(len(datasets_names) / cols)
fig, axs = plt.subplots(nrows=rows, ncols=cols, squeeze=False, figsize=(10, rows * 4))
for ax, dataset_name in zip(axs.ravel(), datasets_names):
for model_idx, model_name in enumerate(model_names):
display = RocCurveDisplay.from_predictions(
y_true[dataset_name],
y_score[model_name][dataset_name],
pos_label=pos_label,
name=model_name,
ax=ax,
plot_chance_level=(model_idx == len(model_names) - 1),
chance_level_kw={"linestyle": ":"},
)
ax.set_title(dataset_name)
_ = plt.tight_layout(pad=2.0) # spacing between subplots
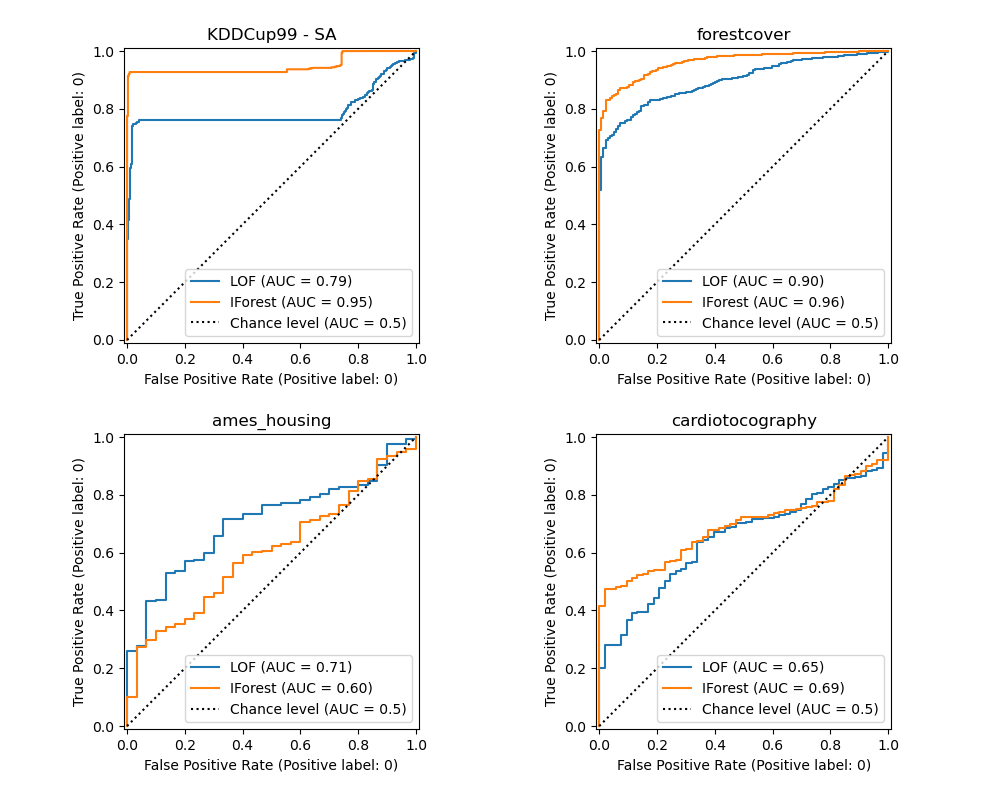
Мы наблюдаем, что после настройки количества соседей LOF и IForest работают схожим образом с точки зрения ROC AUC для наборов данных forestcover и cardiotocography. Оценка IForest немного лучше для набора данных SA, а LOF работает значительно лучше на наборе данных Ames housing, чем IForest.
Однако помните, что Isolation Forest обычно обучается намного быстрее, чем LOF, на наборах данных с большим количеством образцов. LOF необходимо вычислять попарные расстояния для поиска ближайших соседей, что имеет квадратичную сложность относительно количества наблюдений. Это может сделать этот метод непрактичным на больших наборах данных.
Абляционное исследование#
В этом разделе мы исследуем влияние гиперпараметра n_neighbors и
выбор масштабирования числовых переменных в модели LOF. Здесь мы используем Forest covertypes набор данных, так как бинарно закодированные категории вводят естественную шкалу евклидовых расстояний между 0 и 1. Затем нам нужен метод масштабирования, чтобы избежать привилегий для небинарных признаков и который достаточно устойчив к выбросам, чтобы задача их поиска не стала слишком сложной.
X = X_forestcover
y = y_true["forestcover"]
n_samples = X.shape[0]
n_neighbors_list = (n_samples * np.array([0.2, 0.02, 0.01, 0.001])).astype(np.int32)
model = make_pipeline(RobustScaler(), LocalOutlierFactor())
linestyles = ["solid", "dashed", "dashdot", ":", (5, (10, 3))]
fig, ax = plt.subplots()
for model_idx, (linestyle, n_neighbors) in enumerate(zip(linestyles, n_neighbors_list)):
model.set_params(localoutlierfactor__n_neighbors=n_neighbors)
model.fit(X)
y_score = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_score,
pos_label=pos_label,
name=f"n_neighbors = {n_neighbors}",
ax=ax,
plot_chance_level=(model_idx == len(n_neighbors_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
curve_kwargs=dict(linestyle=linestyle, linewidth=2),
)
_ = ax.set_title("RobustScaler with varying n_neighbors\non forestcover dataset")
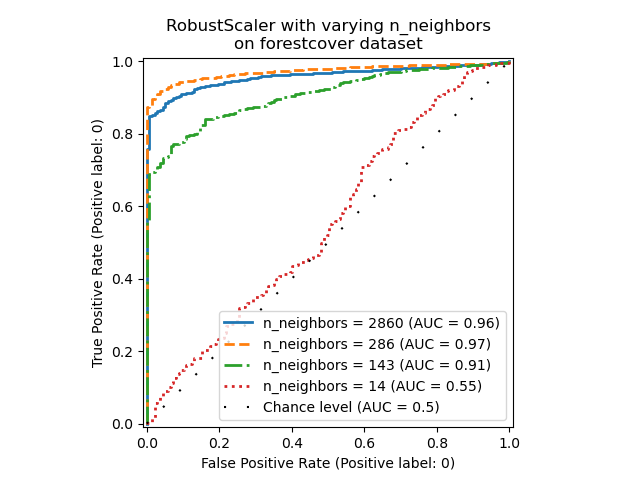
Мы наблюдаем, что количество соседей сильно влияет на производительность модели. Если доступны (хотя бы некоторые) истинные метки, важно настроить n_neighbors соответственно. Удобный способ сделать это — исследовать значения для n_neighbors порядка величины ожидаемого
загрязнения.
from sklearn.preprocessing import MinMaxScaler, SplineTransformer, StandardScaler
preprocessor_list = [
None,
RobustScaler(),
StandardScaler(),
MinMaxScaler(),
SplineTransformer(),
]
expected_anomaly_fraction = 0.02
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_score = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_score,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
curve_kwargs=dict(linestyle=linestyle, linewidth=2),
)
_ = ax.set_title("Fixed n_neighbors with varying preprocessing\non forestcover dataset")
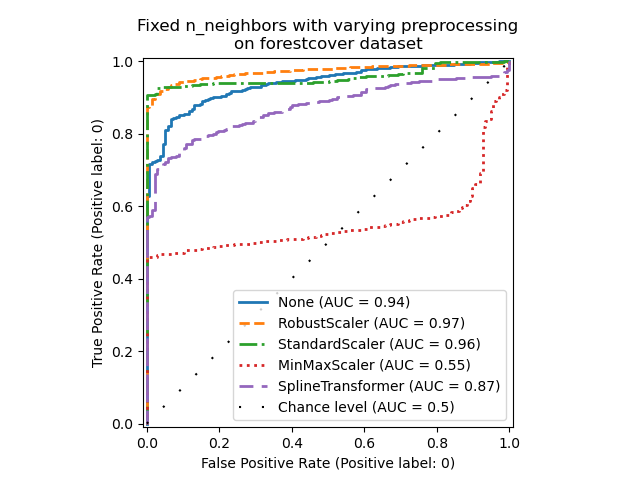
С одной стороны, RobustScaler масштабирует каждый признак независимо, используя по умолчанию межквартильный размах (IQR), который представляет собой диапазон между 25-м и 75-м процентилями данных. Он центрирует данные, вычитая медиану, а затем масштабирует их, деля на IQR. IQR устойчив к выбросам: медиана и межквартильный размах меньше подвержены влиянию экстремальных значений, чем диапазон, среднее значение и стандартное отклонение. Кроме того, RobustScaler не
сжимает маргинальные выбросы, в отличие от
StandardScaler.
С другой стороны, MinMaxScaler масштабирует каждый признак индивидуально таким образом, чтобы его диапазон отображался в диапазон от нуля до единицы. Если в данных присутствуют выбросы, они могут сместить распределение в сторону минимальных или максимальных значений, что приведет к совершенно другому распределению данных с большими маргинальными выбросами: все невыбросные значения могут оказаться почти сгруппированными вместе в результате.
Мы также оценили отсутствие предобработки вообще (путем передачи None в конвейер),
StandardScaler и
SplineTransformer. Пожалуйста, обратитесь к их соответствующей документации для получения более подробной информации.
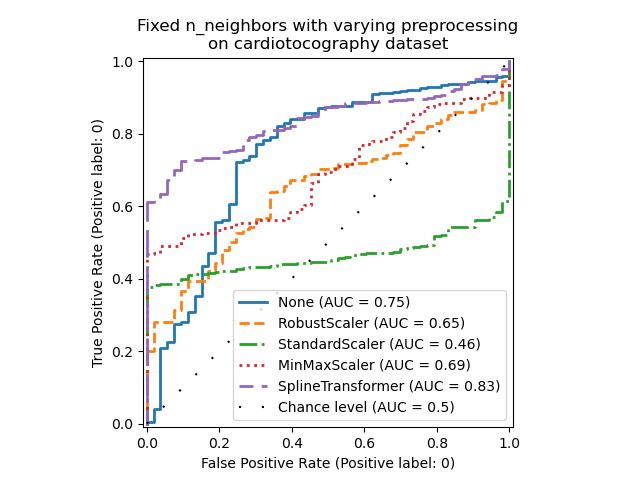
Обратите внимание, что оптимальная предобработка зависит от набора данных, как показано ниже:
X = X_cardiotocography
y = y_true["cardiotocography"]
n_samples, expected_anomaly_fraction = X.shape[0], 0.025
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_score = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_score,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
curve_kwargs=dict(linestyle=linestyle, linewidth=2),
)
ax.set_title(
"Fixed n_neighbors with varying preprocessing\non cardiotocography dataset"
)
plt.show()
Общее время выполнения скрипта: (0 минут 56.835 секунд)
Связанные примеры
Обнаружение выбросов с помощью фактора локальных выбросов (LOF)
Сравнение алгоритмов обнаружения аномалий для выявления выбросов на игрушечных наборах данных
Сравнение влияния различных масштабировщиков на данные с выбросами

Обнаружение новизны с помощью локального фактора выбросов (LOF)